KinDEL数据集:包含8100万个小分子的库,为激酶抑制剂的发现提供了一个丰富且功能强大的资源。
2024-10-12,在药物发现领域,Insitro公司创建了KinDEL,一个针对两个激酶靶点(MAPK14和DDR1)的大规模、公开可用的DNA编码库数据集。这个数据集不仅推动了计算技术的发展,还通过不同的机器学习技术,为识别潜在药物提供了预测模型,具有重要的科研和临床意义。
一、研究背景:
DNA编码库(DEL)作为一种组合小分子库,在药物发现中扮演着重要角色,它通过高通量筛选对治疗相关靶点的小分子库进行高效筛选。然而,公共DEL数据集的有限可用性限制了计算技术的进一步发展。
目前遇到困难和挑战
1、DEL合成和选择过程中的固有噪声,导致数据存在多种偏差源,需要现代机器学习方法来从数据中学习信号。
2、DEL实验通常测量带有DNA条码的分子的亲和力(即on-DNA结合),而在实际治疗活动中,药物样分子是在没有DNA标签的情况下进行测试的(即off-DNA),这意味着DEL数据受到DNA和分子与其连接的影响。
3、反应产率的不确定性和聚合酶链反应(PCR)扩增过程中的偏差,为过程增加了额外的噪声。
数据集地址:KinDEL|药物发现数据集|机器学习数据集
二、让我们一起来看一下KinDEL数据集
KinDEL(Kinase Inhibitor DNA-Encoded Library)是一个包含8100万个小分子的库,这些分子用于与两个蛋白质靶点MAPK14和DDR1的选择实验。KinDEL数据集的特点是高一致性的实验重复,提供了大量的监督数据,有助于机器学习社区开发解决药物发现中的小分子化学问题的方法。
DEL的构建是一个逐步的过程,每个合成子(synthon)都由DNA标签指定并逐一添加。此外,每个分子不仅有单一的编码,多个DNA编码映射到最终的分子结构,这些冗余编码有助于减少后续DNA测序步骤中的潜在偏差。
研究人员可以使用KinDEL数据集来开发和验证预测模型,通过生物物理实验数据来测试这些模型的有效性。
基准测试 :
研究人员提供了使用生物物理实验数据验证的基准任务,这些任务可以帮助比较不同建模技术在DEL数据上的应用效果。

(上图)DEL 以顺序方式合成;在每一步中,DNA 密码子都指定要连接的下一个构建块(synthon)。(下)完全合成的 DEL 分子的示例,其中构建块和密码子进行了颜色编码以实现可视化。通常,还有一个连接子将分子连接到 DNA,这是一个 6 碳链。

通过将 DEL 与固定在链霉亲和素珠上的目标蛋白靶标相结合来进行 DEL 选择实验。经过多轮洗涤后,将紧密结合物从蛋白质上洗脱出来,并对其相应的 DNA 进行测序以获得计数数据。

KinDEL 数据集中化学性质的分布。这些选定的特性通常用于评估分子的药物相似性。据 Shultz (2018) 报告,浅蓝色区域标记了为 FDA 批准的所有口服新化学实体计算的第 10 个和第 90 个百分位数。QED:药物相似性的定量估计 (Bickerton et al., 2012)。

数据集的 3D 立方体可视化,其中每个轴对应于 DEL 中的不同合成子。图中的点是富集度最高的化合物(使用泊松富集测量)。线性模式可以解释为富集的 disynths,即通常与蛋白质靶标结合的两个 synthon 的组合。
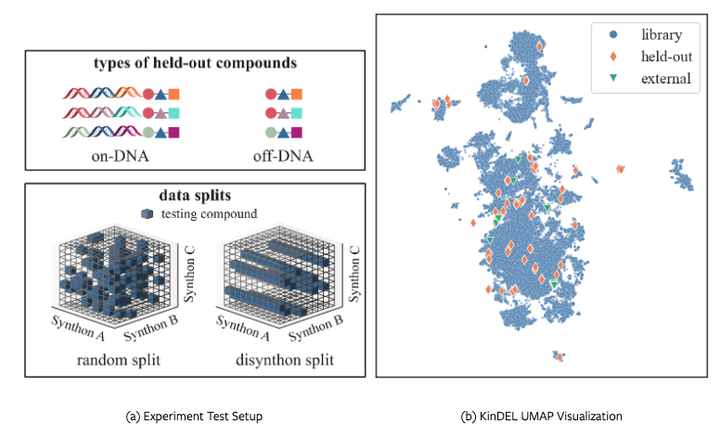
(a) 检测数据准备包括选择用于 DNA 上和脱离 DNA 重组(上)的保留检测化合物以进行结合测定,以及用于准备内部检测集的两种类型的数据拆分(下)。(b) 使用化合物之间的 Tanimoto 距离构建的 KinDEL 的 UMAP 可视化。为保留的测试集选择的化合物被描述为橙色菱形(库内)和绿色三角形(外部)。
三、让我们来看一下KinDEL数据集应用场景:
比如,我是一名药物研发科学家
说起我做药物研发的日子,那真是一把辛酸泪。整体像个老黄牛一样,在实验室里捣鼓各种化合物。你知道的,我们得找到那些能“锁住”特定蛋白质的分子,这样它们就不会在身体里乱来了,也就能够治疗疾病。但是,这就像是在一片黑森林里找一根特定的小草,工作量巨大,还不一定能找到。
比如说,我之前在研究一种治疗癌症的药物。癌症这玩意儿,就是因为细胞里的一些蛋白质太活跃,导致细胞疯狂分裂。我们的任务就是找到能“按住”这些蛋白质的分子。但是,要找到这样的分子,得合成成千上万种化合物,然后一个个测试它们对蛋白质的作用。这不仅耗时耗力,而且成本还特别高。
自从我开始使用KinDEL数据集来训练我的机器学习模型,我的工作就发生改变。
这个数据集包含了8100万个小分子,这些分子都已经被测试过,看它们是否能和特定的激酶蛋白质结合。激酶这种蛋白质,就像是细胞里的一个开关,如果它一直开着,细胞就会出问题。
我给模型输入了大量的数据,它就能学习到哪些分子的形状、大小和电荷等特点,是和蛋白质结合的关键。
现在,我只需要把新的化合物输入模型,它就能预测这些化合物是否有可能成为有效的药物。这样,我就可以把精力集中在那些最有可能成功的化合物上,而不是盲目地一个一个测试。
这个改变对我来说简直是翻天覆地的。以前我可能要花上几个月甚至几年的时间来筛选化合物,现在可能只需要几周,甚至几天。而且,因为模型是基于大量数据训练的,它的预测准确性也大大提高。这不仅让我的工作更有效率,也让我对找到新药物更有信心了。
