m6A-BERT——基于 BERT 的模型可用于预测具有遗传信息的 MRNA 的功能
1. 导言
1.1 关于 mRNA
人类和其他生物的遗传信息都包含在DNA中,但要根据遗传信息实际控制我们的身体功能,必须先将 DNA 中的遗传信息复制到一种叫做 mRNA 的物质上,然后利用 mRNA 信息合成蛋白质。
换句话说,众所周知,DNA 中的信息需要复制到另一种介质mRNA 中,才能转化为负责人体功能的蛋白质;DNA、mRNA 和蛋白质之间的关系可以这样比喻:DNA 是菜谱,mRNA 是菜谱的副本、如果我们把蛋白质看作一道成品菜,就更容易理解了。
1.2 关于 mRNA 功能的调控
有时,mRNA 会进行一些操作,在 mRNA 上添加一些装饰性修饰,这些修饰被称为 “修饰”。这种修饰的一个典型例子是m6A修饰,它表示在 "A "的氮原子上添加了一个 “甲基”。m6A 是一个字母,已知有**“A”、“G”、"C "和 “U”**,因为构成 mRNA 字符串的有四个不同的单元。
众所周知,mRNA 经过这种修饰后会吸引负责调节自身降解的蛋白质。然而,m6A修饰并不一定会导致mRNA降解,其详细机制尚未完全阐明。
这种稳定性的调节与各种细胞和生物过程(包括急性髓性白血病中的****癌症干细胞)高度相关,人们一直期待着对其进行阐明。
1.3 研究背景
因此,本研究建立了一个名为 m6A-BERT的模型,用于预测具有m6A修饰的 mRNA 是否会降解。此外,利用mRNA的寿命数据(半衰期),他们还提出了m6A-BERT-Deg,一个利用微调技术对该模型进行改进的版本。
mRNA的半衰期与mRNA 的降解速度有关,是了解降解机制的一个重要参数。
与其他最先进的基于深度学习的方法相比,m6A-BERT-Deg的高精确度证实了它的有效性。
2.模型结构
2.1 整体模型
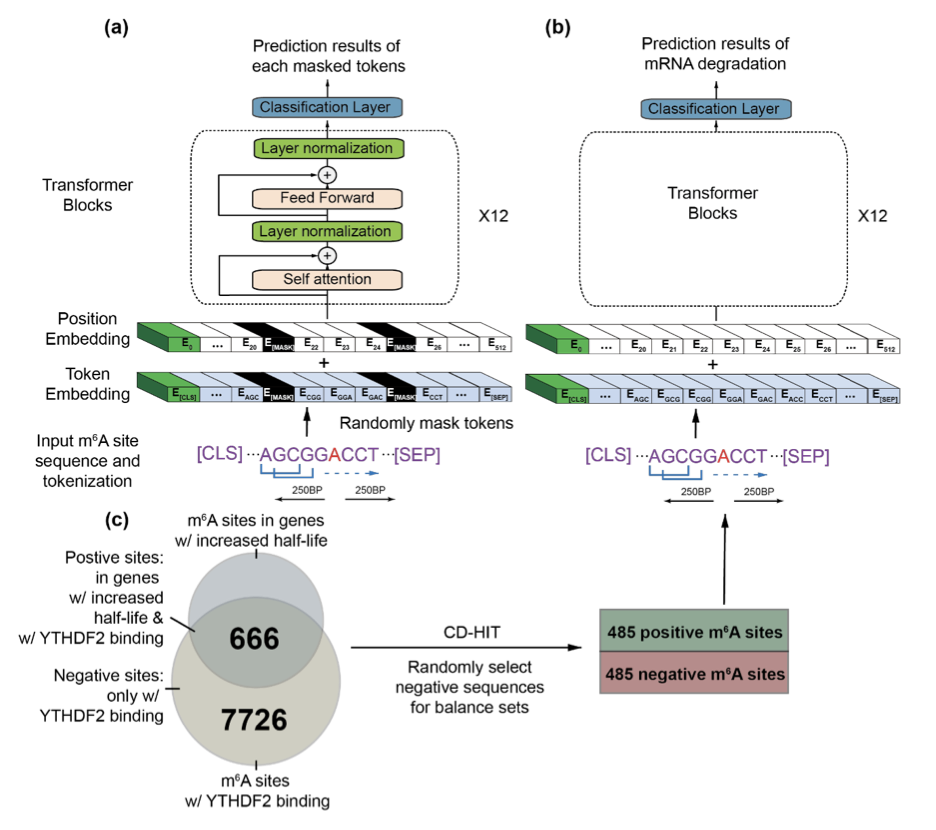
mRNA 的结构就像一条细长的链条,四种组成单位分别连接在一起,形成一个字符串。这意味着,如果每个成分都用一个字母缩写来表示,那么 mRNA 就可以用字符串的形式来表示�
m6A-BERT基于我们熟悉的自然语言处理****BERT模型,其整体情况如上图所示,包括 A 所示的预训练和 B 所示的使用 C 数据(下行)进行微调的过程。
模型各结构的详情如下。
2.2 细节
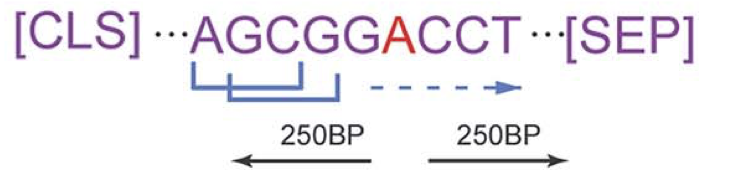
在该模型中,mRNA字符序列被用作标记化的输入数据。如图所示,标记化是使用滑动窗口技术进行的。
这种技术的基础是提取具有一定宽度和长度的 mRNA 序列的一部分(上图中的长度设置为 3),然后通过逐渐移动要提取的部分与整个字符串的相对位置,对其进行标记化。
在这里,三个字符串的集合被视为一个单一的块。换句话说,在图中,“AGC”(第一个至第三个字母)被视为单个标记。此外,“GCG”(对应第二至第四个字母)、“CGG”、"GGA "等也分别被视为标记。
请注意,图中红色区域指的是实际进行 m6A 修改的区域,列中距离该区域250 个字符以内的数据都包含在分析中。标记**[CLS]和[SEP]分别指在开头和结尾添加的特殊标记**。
在上面的例子中,我们考虑了宽度为3的情况,但在本文中,我们将宽度设为 4,即 3、4、5 和 6,并以不同的粒度进行预训练(本文中的实验表明,在这些宽度下,准确率几乎相同)。
2.3 初步研究细节

在预训练过程中,m6A 序列中15%的标记会被随机屏蔽。也就是说,这些标记会被替换成**[MASK]标记**,如图中黑色所示。
然后,由 12 层组成的变换器模块和分类层通过嵌入得到的输出来预测掩码标记。
请注意,预研究期间使用了数据集 m6A-AtlasV2,该数据集显示了 24 种组织和细胞系中含有 m6A 修饰的 mRNA 序列。
2.4 更多关于微调
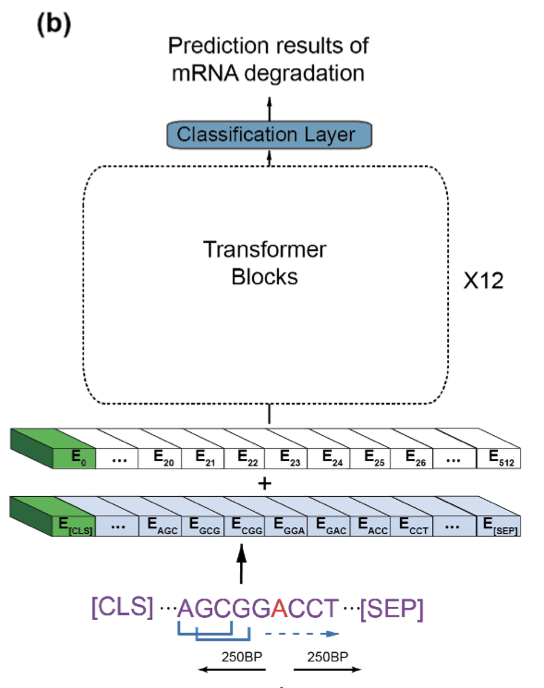
在微调过程中,预训练模型会引入一个二进制分类层。如果该层预测到 mRNA 降解有调节作用,则输出1;如果预测到没有调节作用,则输出0。
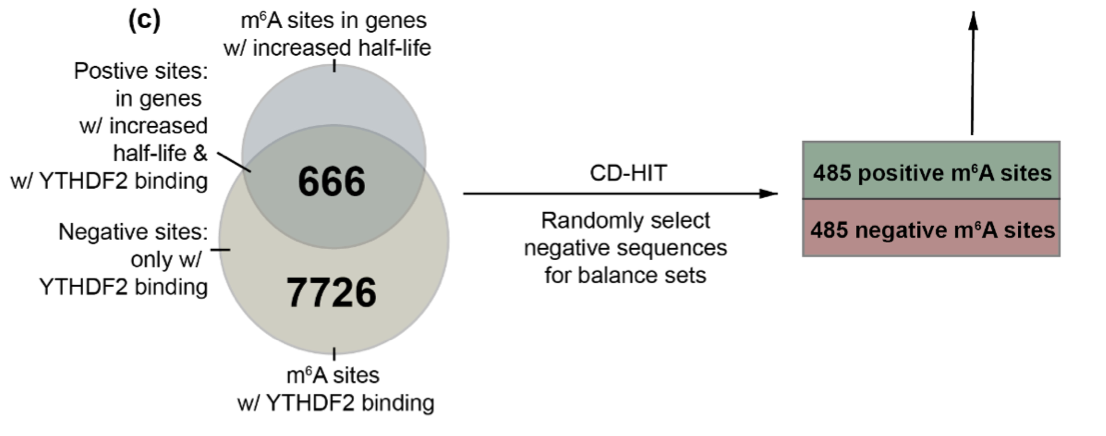
在微调过程中,数据集的构建如上图所示。上面的圆圈表示 "mRNA 经 m6A 修饰且半衰期延长"的位点数量,下面稍大的圆圈表示 "mRNA 经****m6A 修饰且与某种蛋白质(正式名称为 YTHDF2)结合****导致基因降解"的位点数量。
数据集还从属于下圆但不属于上圆(即蛋白质被束缚且半衰期不延长)的 7726 个位点中随机选取 485 个位点作为负集,从属于两个圆(蛋白质被束缚且半衰期延长)的位点中随机选取 485 个位点作为正集,从属于两个圆(即蛋白质被束缚且半衰期延长)的 7726 个位点中随机选取 485 个位点作为正集。数据集是从两个圆圈内(即蛋白质被结合且半衰期延长)的 485 个位点中随机抽取的 485 个位点组成的正集。
换句话说,在诱导 mRNA 降解的蛋白质与m6A 的修饰位置结合的数据中,实际发生降解的情况被视为阳性数据集,而没有发生降解的情况被视为阴性数据集。
3. 项目部署
3.1. 环境设置
建议在设置环境时使用 Conda,pytch = 1.10.2,captum = 0.5.0,python = 3.6。
git clone https://github.com/TingheZhang/m6A-BERT.git
cd m6A-BERT
conda create --name m6A-BERT python==3.6
activate m6A-BERT
3.2 数据处理
在进行微调和预测之前,应该将输入序列处理成 K-mers:
-
通过导出变量设置所需的 K-mer 长度(K) :
export KMER=3 ### Select K from 3 to 6 -
指定原始数据的路径和保存已处理数据的位置:
export RAW_DATA_PATH=YOUR_RAW_DATA_PATH export DATA_PATH=THE_PATH_YOU_WANT_TO_SAVE_PROCESS_DATA -
为数据平衡设置一个种子编号:
export SEED=452 ### Choose your desired seed number -
在 Linux bash 环境中运行以下命令以获得正确的输入:
python3 get_input.py --do_val --kmer $KMER --extend_len 250 --task finetune --data_dir $RAW_DATA_PATH --save_dir $DATA_PATH --seed $SEED确保在 RAW _ DATA _ PATH 下,正序列在 pos.fa 中,负序列在 neg.fa 中。
3.3. 微调模型
M6A-BERT 可以很容易地为下游分析进行微调,代码可以简单地在 Linux bash 中运行:
export KMER=3 ### select the K from 3 to 6, have to match the K in the data process section
export RAW_DATA_PATH= YOUR_RAW_DATA_PATH
export DATA_PATH=THE_PATH_YOU_SAVED_PROCESSED_DATA
export MODEL_PATH=THE_PATH_OF_PRETRAINED_MODEL
export OUTPUT_PATH=THE_PATH_TO_SAVE_FINETUNED_MODEL
python3 run_finetune_degradation.py --model_type dna --tokenizer_name=dna$KMER --model_name_or_path $MODEL_PATH
--task_name dnaprom --do_train --do_eval --data_dir $DATA_PATH --save_steps 50 --logging_steps 50
--max_seq_length 512 --per_gpu_eval_batch_size=40 --per_gpu_train_batch_size=40 --learning_rate 1e-6 --num_train_epochs 100
--output_dir $OUTPUT_PATH --n_process 10 --hidden_dropout_prob 0.1 --evaluate_during_training --weight_decay 0.01
请根据图形处理器的内存大小调整每 GPU _ eval _ batch _ size 和每 GPU _ train _ batch _ size。
预先训练 m6A-BERT 可以从这里下载我们的微调 m6A-BERT-DEG 可以从这里下载
3.4. 做出预测
经过微调并获得 m6A-BERT-DEG 模型,人们可以通过使用以下推荐信进行预测:
export KMER=3 ### select the K from 3 to 6, have to match the K in the data process section
export RAW_DATA_PATH= YOUR_RAW_DATA_PATH
export DATA_PATH=THE_PATH_YOU_SAVED_PROCESSED_DATA
export MODEL_PATH=THE_PATH_OF_PRETRAINED_MODEL
export OUTPUT_PATH=THE_PATH_TO_SAVE_FINETUNED_MODEL
export FINUE_TUNED_MODEL_PATH=$OUTPUT_PATH/checkpoint-# ##change # to user selected numbers
python3 run_predict_degradation.py --model_type dna --tokenizer_name=dna$KMER --model_name_or_path $MODEL_PATH
--task_name dnaprom --data_dir $DATA_PATH --save_steps 50 --logging_steps 50 --do_predict
--max_seq_length 512 --per_gpu_eval_batch_size=50 --per_gpu_train_batch_size=50 --learning_rate 1e-6 --num_train_epochs 100
--output_dir $FINUE_TUNED_MODEL_PATH --n_process 1 --evaluate_during_training --predict_dir $OUTPUT_PATH
3.5. 可视化
经过微调并获得 m6A-BERT-DEG 模型,人们可以通过使用以下推荐信进行预测:
export KMER=3 ### select the K from 3 to 6, have to match the K in the data process section
#export RAW_DATA_PATH= YOUR_RAW_DATA_PATH
export DATA_PATH=THE_PATH_YOU_SAVED_PROCESSED_DATA
export FINUE_TUNED_MODEL_PATH=THE_PATH_OF_FINUE_TUNED_MODEL
export MODEL_PATH=THE_PATH_OF_PRETRAINED_MODEL
export OUTPUT_PATH=THE_PATH_TO_SAVE_OUTPUT
export MOTIF_PATH=THE_PATH_TO_SAVE_MOTIF
export DATASET=dev ##select the dataset to visulize, could be train, dev, test
export TASK=attr ##select the visulzation methods, could be attn (attention weights) or attr (attribution scores)
python3 visualize_all.py --kmer $KMER --model_name_or_path $MODEL_PATH --model_path $FINUE_TUNED_MODEL_PATH --output_dir $OUTPUT_PATH
--data_dir $DATA_PATH --data_name $DATASET --vis_task $TASK --batch_size 50
4. 实验
4.1 关于模型的评估指标
我们选择了五个指数来评估模型的性能:ACC、马修斯相关系数、AUC、准确度和可重复性。马修斯相关系数是二元分类问题中使用的评估指标之一,用于评估模型在不平衡数据集中的性能。请注意,模型的性能是通过五部分交叉验证法进行比较的。
4.2 实验结果
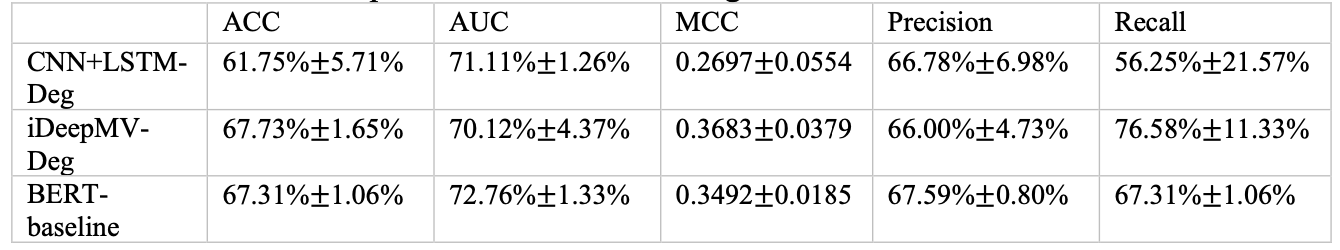
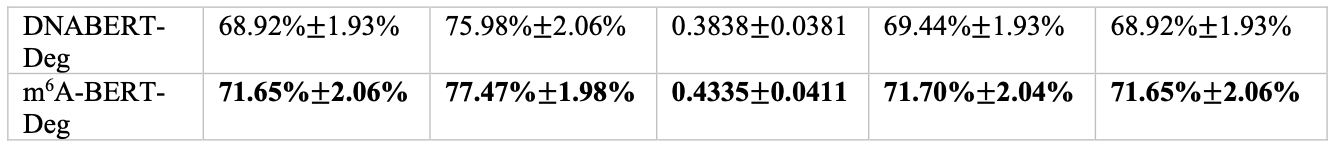
与基线模型相比,m6A-BERT-Deg的预测性能如表所示
在本文中,为了证明先验学习的效果,比较了未经先验学习训练的BERT 基准、DNABERT-Deg(其中使用本文介绍的方法对传统方法 DNABERT 进行了微调)以及iDeepMVDeg 和****CNN+****LSTM-Deg的预测性能。LSTM-Deg作为传统模型进行比较,以验证预测性能。
实验结果表明,m6A-BERT-Deg在所有模型中表现最佳。特别是,与没有先验学习的方法相比,ACC 和 AUC 提高了约 4%,表明了先验学习的有效性。
此外,m6A-BERT-Deg还被用于验证 HEK293T 细胞系(细胞系是指在体外持续生长的细胞群)对 mRNA 降解的调控,并与另一种测序方法m6A-express 的结果进行了比较。论文显示,该模型的预测结果与另一种序列分析方法 m6A-express 的结果相比是正确的。
4.3 Token贡献分提供的考虑因素
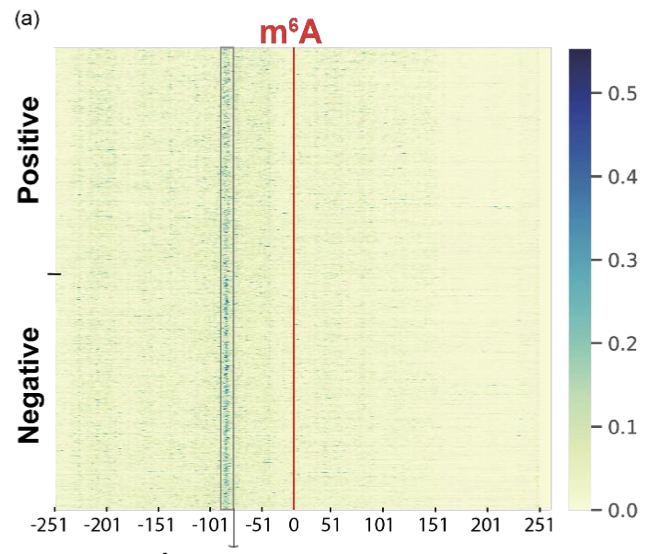
如图所示,作者绘制了一张热图,用颜色强度来直观显示归因分数的大小。归因分数是衡量每个标记对预测的贡献程度,分数越高,表示标记对预测的影响越大。
请注意,该图的上半部分显示的是正向数据集的归因得分,而下半部分显示的是负向数据集的归因得分。横轴显示的是字母序列中发生 m6A 修饰的部分,可以肯定的是,数字对应的是距离发生 m6A 修饰部分的字母数。
从图中可以看出,相对于横轴在-100 左右的区域,有些区域的分数部分较高(蓝点散布在周围)。
从图中可以看出,m6A 修饰本身所在区域的归因分值较低(即标记对学习的贡献****较低),而在 m6A 位点的上游(即负)区域,归因分值较高(即标记对学习的贡献较高)。研究结果。
这表明,发生修饰的上游区域可能对 mRNA 降解的调控有重要影响。
4.4 阐明新生物机制的潜力
此外,论文还验证了哪些蛋白质更频繁地与 RNA 的某些序列结合。作者指出,其中一些蛋白质能促进 mRNA 的稳定性,并明确指出,这可能揭示了一种新的生物学机制,通过这种机制,mRNA的稳定性可能会因为mRNA 降解被阻止而得到增强。报告还指出,一种新的生物机制可能已经被阐明。
5. 总结
本研究提出了基于 BERT 的****m6A-BERT-Deg模型,用于预测 mRNA 因 m6A 修饰而降解的情况。
该模型的训练方法是**:将mRNA 序列标记为字符串,进行预训练以预测屏蔽标记,然后进行微调**,引入二元分类层以预测分解。
该模型的性能高于其他未经预训练的先进模型和以往的模型。在真实细胞上进行的实验也证实了该模型的准确性。
使用基于标记贡献的归因得分进行的进一步分析表明,m6A 修饰位点上游的得分较高,表明该区域对调节 mRNA 降解很重要。
BERT 模型是能够通过考虑这些嵌入层的贡献来充分考虑生物背景知识的最佳部分,我认为我们现在能够通过这种考虑来阐明新的机制是非常了不起的。
