大语言模型LLMs在医学领域的最新进展总结
我是娜姐 @迪娜学姐 ,一个SCI医学期刊编辑,探索用AI工具提效论文写作和发表。

相比其他学科,医学+AI,是发表学术成果最多的领域。
医学数据的多样性和复杂性(包括文本、图像、基因组数据等),使得传统的数据分析方法难以全面整合和解析。
而大语言模型,特别是多模态模型,可以综合分析不同类型的数据,建立跨模态关联,提供从文本到影像的深度理解。
这篇文章,娜姐用主题检索的方式,总结了近一年来,医学领域大模型的研究进展。主要分为辅助诊断、药物研发、基因组学、医患沟通等方面。
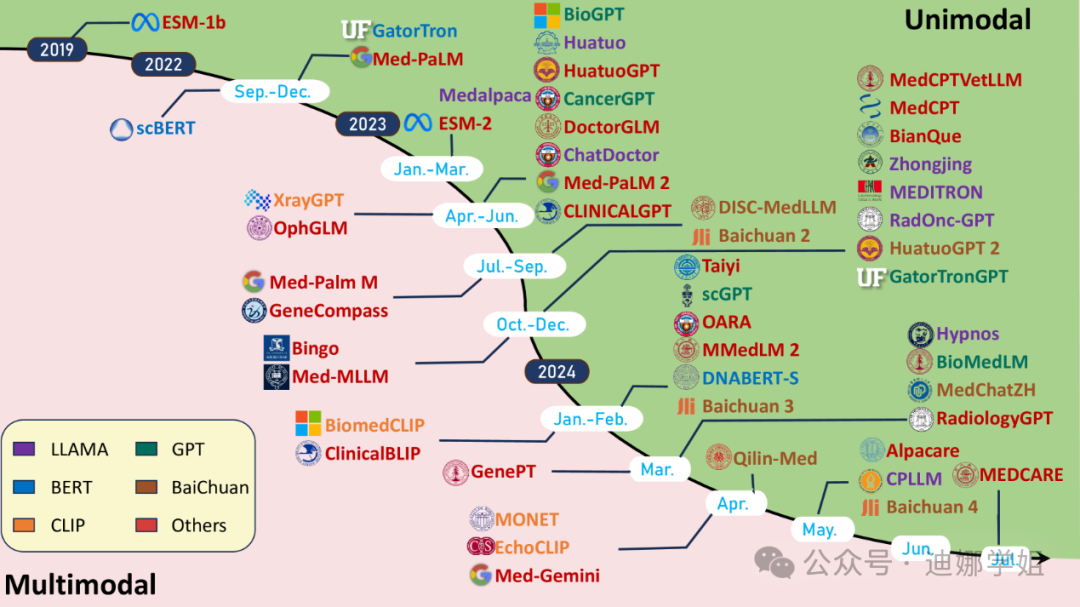
图1 生物医学领域LLMs的发展概览。从左到右,展示了单模态(绿色部分)和多模态(粉色部分)模型随时间的研究进展【1】。
一、LLM在医学细分领域的研究进展:
1 医学影像辅助诊断:
哈佛大学生物医学信息学助理教授余坤兴2024年9月发表在Nature上的成果【2】,他们团队开发了一款临床组织病理学成像评估基础模型(CHIEF,Clinical Histopathology Imaging Evaluation Foundation)。该CHIEF模型能够对源于肺、乳腺、前列腺、结直肠、胃、食道、肾、脑、肝、甲状腺、胰腺、宫颈、子宫、卵巢、睾丸、皮肤、软组织、肾上腺和膀胱等组织的19 种癌症进行诊断,检测准确率接近 94%。
团队正在与业界合作,希望将 CHIEF 模型发展为临床辅助诊断工具,并在准备 FDA 的相关审批工作。
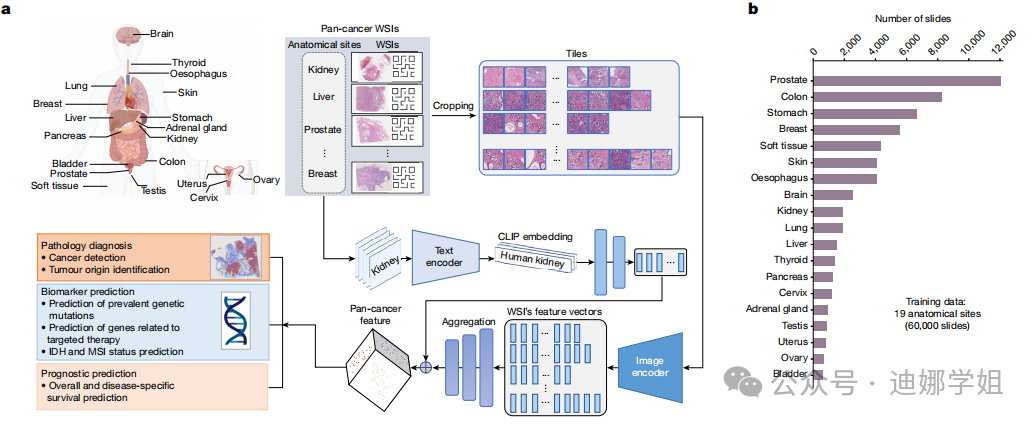
图2 CHIEF 模型概述(来源:Nature,【2】)
美国商业公司Paige研发的病理学基础模型Virchow,能够实现泛癌症检测,在九种常见癌症和七种罕见癌症中,样本级接收者操作特征曲线下面积达到 0.95。此外,在训练数据较少的情况下,Virchow也能够实现与生产中的组织特异性临床级模型相似的性能,并在某些罕见癌症变种上超越它们【3】。
哈佛大学医学院研究团队近期开发了一个用于人类病理学切片鉴定的视觉语言通用 AI 助手——PathChat。该系统通过自我监督学习对来自 100 万多张切片的图像片段进行预训练,能够从活检切片中正确识别疾病,准确率近 90%,超越GPT-4V。
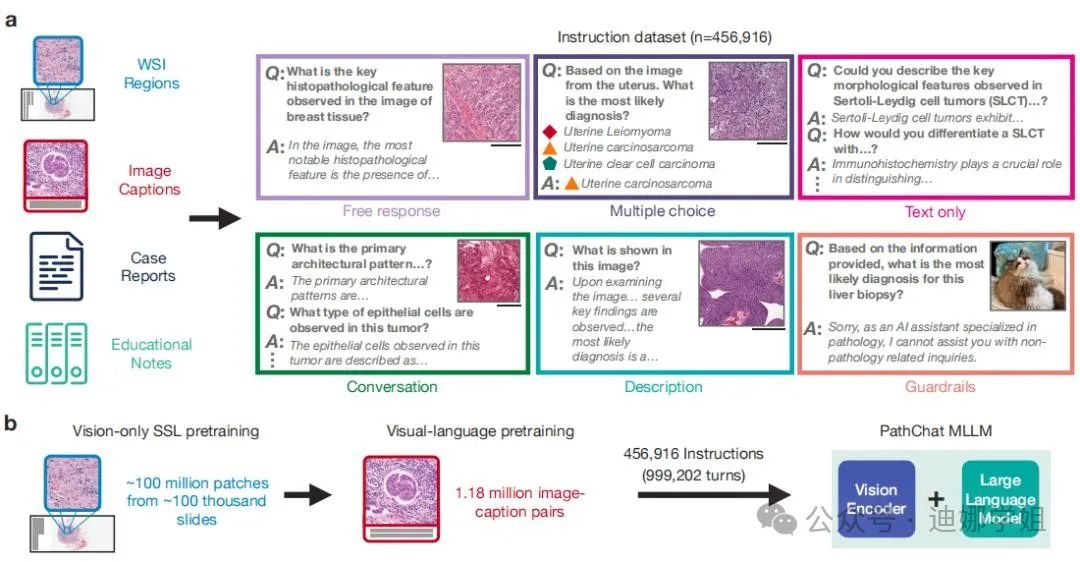
图3 PathChat 的训练和构建过程。(来源:Nature,【4】)
SkinGPT-4,是一个基于多模态大型语言模型的互动皮肤病诊断系统。基于Llama-2-13b-chat大型语言模型,通过52,929 张病理图像及临床概念进行训练。用户可以上传自己的皮肤照片进行诊断。系统自主评估图像,识别皮肤状况的特征和类别,进行深入分析,并提供互动治疗建议【5】。
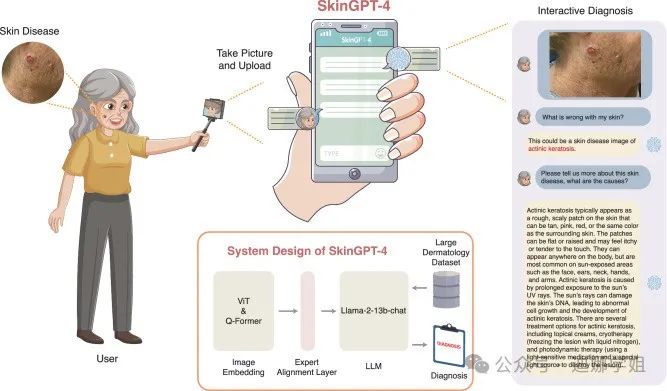
图4 SkinGPT-4 是一个基于多模态大型语言模型的互动皮肤病诊断系统。(来源:Nature Communications,【5】)
中山大学附属第一医院针对甲状腺结节影像及病理评估的一项实验表明,725 名患者中的 1161 幅甲状腺结节的影像诊断对比,ChatGPT 4.0 和 Bard 显示出显著到几乎完美的内部一致性,与两名高级影像师和一名初级影像师的人机交互策略相当,并超过了仅有一名初级影像师的人机交互策略【6】。
中国学者开发的肺尘病诊断大模型PneumoLLM,开辟了针对数据稀缺的职业病应用LLMs的新范式,通过广泛的实验展示了大模型在诊断尘肺病方面的优越性【7】。
2 药物开发
浙江大学人工智能医学创新研究院开发了LEDAP模型,利用了基于LLM的生物文本特征编码来预测药物-疾病关联、药物-药物相互作用和药物-副作用关联。LEDAP 在与其他流行的 DBA 分析工具相比时展示了其显著的竞争力【8】。
哈佛医学院研究人员开发的TxGNN 模型,在涵盖 17,080 种疾病的疾病机制和 7,957 种药物的作用机制的医学知识图谱上进行训练,旨在解决现有药物的新应用,为治疗选择有限且分子数据稀缺的疾病识别候选药物【9】。
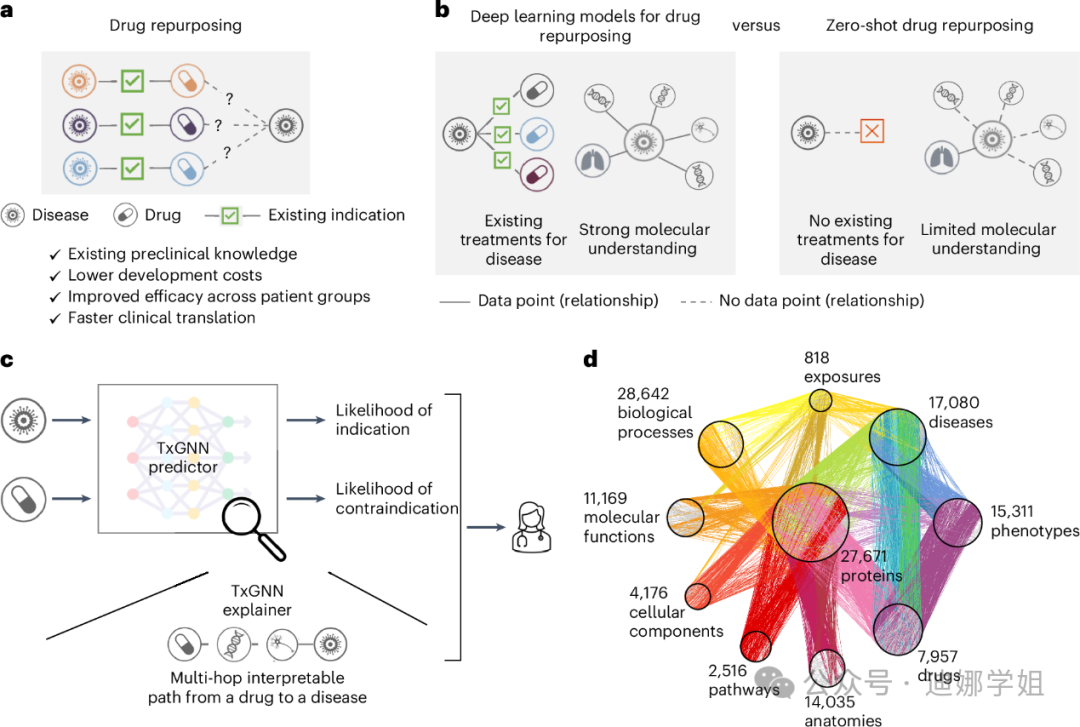
图5 TxGNN:该几何深度学习模型结合了一个庞大而全面的生物知识图谱,以准确预测任何给定疾病-药物对的适应症或禁忌症的可能性,适用于老药新用途的开发。(来源:Nature Medicine【9】)
中国科学技术大学联合微软研究院,开发了 TamGen--一种采用类似 GPT 的化学语言模型的方法,能够实现靶向感知的分子生成和化合物精炼。将 TamGen 集成到药物发现流程中,并识别出 14 种对结核病 ClpP 蛋白酶表现出显著抑制活性的化合物,其中最有效的化合物的半最大抑制浓度(IC50)为 1.9 μM【10】。
理解化学干扰的转录响应对于药物发现至关重要。中科院计算技术研究所联合合作者,开发了PRnet深度生成模型,能够预测从未在大规模和单细胞水平上进行实验干扰的新化学扰动的转录响应(transcriptional response)。PRnet 使基因水平的响应解释和基于基因特征的计算药物筛选成为可能。PRnet 生成了一个大规模的扰动特征整合图谱,涵盖 88 个细胞系、52 种组织和各种化合物库。并成功推荐了 233 种疾病的药物候选者【11】。
化疗和靶向治疗中,药物耐药性是一个关键挑战。佛罗里达大学团队提出的DrugFormer 模型,整合了序列化基因标记和基于基因的知识图谱,以高精度预测单细胞水平的药物耐药性。来自不同癌症类型的全面单细胞数据分析突显了 DrugFormer 在识别耐药细胞和揭示潜在分子机制方面的有效性【12】。
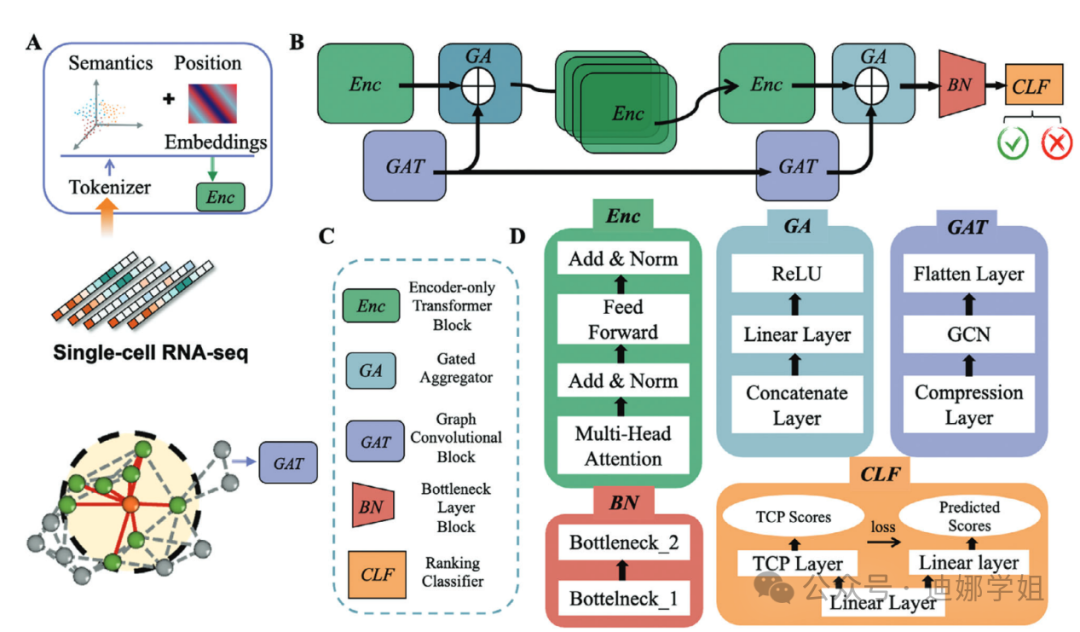
图6 DrugFormer 模型的整体框架。(图源:Advanced science)
3 基因组学
布朗大学团队开发了多模态深度学习模型 EPBDxDNABERT-2。使用包含 690 个 ChIP-seq 实验结果的染色质免疫沉淀测序(ChIP-Seq)数据进行训练, EPBDxDNABERT-2 显著提高了 660 多个 TF-DNA 的预测,揭示了在全基因组关联研究中发现的与疾病相关的非编码变异的机制【13】。
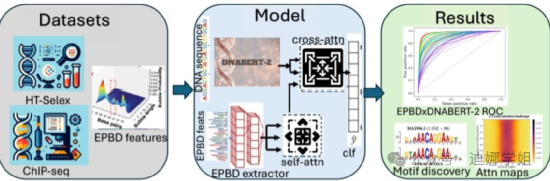
图7 EPBDxDNABERT-2的构建过程。
受大型语言模型的启发,北京理工大学邵斌等开发了一种用于基因组的长上下文生成模型megaDNA。模型的基础能力,包括预测必需基因、遗传变异效应、调控元件活性以及未注释序列的分类。此外,它能够生成长度达到 96 K 碱基对的 de novo 序列,这些序列包含潜在的调控元件和具有噬菌体相关功能的注释蛋白【14】。该生成基因组模型代表了全功能基因组从零开始设计的第一步。
Memorial Sloan Kettering癌症中心报告了一种基于遗传,而非组织病理学数据训练的人工智能算法的构建,该算法能够准确分类浸润性乳腺癌(ILCs)并揭示 CDH1 失活机制,为开发应用于全切片图像的诊断人工智能模型提供了正交真实数据利用的基础。揭示了与强基因型-表型相关性相关的遗传改变可用于开发应用于病理学的人工智能系统,从而促进癌症诊断和生物学发现【15】。
肽在许多生物活动中发挥着关键作用,是药物设计中有前景的候选者。然而,准确预测蛋白质-肽结合亲和力仍是一项挑战。针对这一问题,北京工业大学团队开发了一种基于卷积神经网络和多头注意力的预测模型 PepPAP,该模型仅依赖于序列特征。PepPAP可用于广泛基因组蛋白-肽结合亲和力预测,并有潜力为基于肽的药物设计提供有价值的见解【16】。
4 其他
对于肌萎缩侧索硬化症(ALS)患者来说,眼动追踪技术使用户能够利用键盘,输入文本以进行语音输出和电子消息传递。但是效率仍远低于语言交流。谷歌及合作者团队利用微调的LLMs和对话上下文,开发了一种名为 SpeakFaster 的文本输入用户界面,将高度缩写的英语文本扩展为所需的完整短语,具有非常高的准确性。与传统基线相比,文本输入速度显著提高(29-60%)并节省了运动动作【17】。
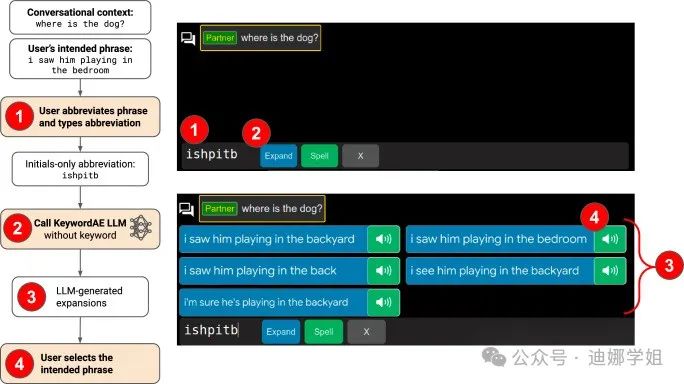
图8 SpeakFaster 用户界面。(来源:Nature Communications 【17】)
医患沟通
2024年7月,中国医学科学院基础医学研究所龙尔平团队与耶鲁大学陈庆宇合作,基于35418例真实导诊对话信息形成的知识库作为训练数据,构建了SSPEC导诊大模型【18】。相比人类导诊,SSPEC在事实性、安全性、共情能力均展现出明显优势,在真实应用场景中,降低了11.2%的重复沟通和5.4%的医患冲突比例。
二、LLM在医疗领域的优势总结
-
LLM 在医学中带来了更准确的诊断和预测,促进了早期疾病检测和个性化治疗计划的制定。
-
为医生提供实时决策支持和更新的医学知识,从而优化临床决策过程。
-
个性化治疗方案并促进药物开发,为患者提供更精确的治疗选择和用药选择。
-
LLM有潜力增强患者管理和医疗流程,提高医疗效率和患者护理质量。
-
LLM可以支持医学教育和医疗知识的传播,促进医学生和从业者的持续学习和改进。
三、挑战:
在生物医学研究中整合LLM带来了机遇,同时也提出了重要的伦理考虑。包括:
潜在的算法偏见;
在人工智能辅助的临床决策中的知情同意;
医疗责任和法律责任问题;
对数据所有权和隐私的担忧。
应对这些挑战需要人工智能研究人员、医疗专业人员、伦理学家和政策制定者之间的合作,以制定稳健的指导方针和监管框架。
总的来说,LLM正成为医学创新的新引擎。ChatGPT等通用模型已展现出卓越的推理和分析洞察能力,随着垂直领域模型的不断深化,未来智能助手将切实融入医生的日常实践,为诊疗质量和效率的提升提供有力支撑。
参考文献:
1 Wang, C., Li, M., He, J., Wang, Z., Darzi, E., Chen, Z., Ye, J., Li, T., Su, Y., Ke, J., Qu, K., Li, S., Yu, Y., Liò, P., Wang, T., Wang, Y.G., & Shen, Y. (2024). A Survey for Large Language Models in Biomedicine. ArXiv, abs/2409.00133.
2 Wang, X., Zhao, J., Marostica, E. et al. A pathology foundation model for cancer diagnosis and prognosis prediction. Nature (2024). https://doi.org/10.1038/s41586-024-07894-z
3 Vorontsov, E., Bozkurt, A., Casson, A., Shaikovski, G., Zelechowski, M., Severson, K., Zimmermann, E., Hall, J., Tenenholtz, N., Fusi, N., Yang, E., Mathieu, P., van Eck, A., Lee, D., Viret, J., Robert, E., Wang, Y. K., Kunz, J. D., Lee, M. C. H., Bernhard, J. H., … Fuchs, T. J. (2024). A foundation model for clinical-grade computational pathology and rare cancers detection. Nature medicine, 30(10), 2924–2935. https://doi.org/10.1038/s41591-024-03141-0
4 Lu, M. Y., Chen, B., Williamson, D. F. K., Chen, R. J., Zhao, M., Chow, A. K., Ikemura, K., Kim, A., Pouli, D., Patel, A., Soliman, A., Chen, C., Ding, T., Wang, J. J., Gerber, G., Liang, I., Le, L. P., Parwani, A. V., Weishaupt, L. L., & Mahmood, F. (2024). A multimodal generative AI copilot for human pathology. Nature, 634(8033), 466–473. https://doi.org/10.1038/s41586-024-07618-3
5 Zhou, J., He, X., Sun, L., Xu, J., Chen, X., Chu, Y., Zhou, L., Liao, X., Zhang, B., Afvari, S., & Gao, X. (2024). Pre-trained multimodal large language model enhances dermatological diagnosis using SkinGPT-4. Nature communications, 15(1), 5649. https://doi.org/10.1038/s41467-024-50043-3
6 Wu, S. H., Tong, W. J., Li, M. D., Hu, H. T., Lu, X. Z., Huang, Z. R., Lin, X. X., Lu, R. F., Lu, M. D., Chen, L. D., & Wang, W. (2024). Collaborative Enhancement of Consistency and Accuracy in US Diagnosis of Thyroid Nodules Using Large Language Models. Radiology, 310(3), e232255. https://doi.org/10.1148/radiol.232255
7 Song, M., Wang, J., Yu, Z., Wang, J., Yang, L., Lu, Y., Li, B., Wang, X., Wang, X., Huang, Q., Li, Z., Kanellakis, N. I., Liu, J., Wang, J., Wang, B., & Yang, J. (2024). PneumoLLM: Harnessing the power of large language model for pneumoconiosis diagnosis. Medical image analysis, 97, 103248. https://doi.org/10.1016/j.media.2024.103248
8 Zhang, H., Zhou, Y., Zhang, Z., Sun, H., Pan, Z., Mou, M., Zhang, W., Ye, Q., Hou, T., Li, H., Hsieh, C. Y., & Zhu, F. (2024). Large Language Model-Based Natural Language Encoding Could Be All You Need for Drug Biomedical Association Prediction. Analytical chemistry, 10.1021/acs.analchem.4c01793. Advance online publication. https://doi.org/10.1021/acs.analchem.4c01793
9 Huang, K., Chandak, P., Wang, Q. et al. A foundation model for clinician-centered drug repurposing. Nat Med (2024). https://doi.org/10.1038/s41591-024-03233-x
10 Wu, K., Xia, Y., Deng, P., Liu, R., Zhang, Y., Guo, H., Cui, Y., Pei, Q., Wu, L., Xie, S., Chen, S., Lu, X., Hu, S., Wu, J., Chan, C. K., Chen, S., Zhou, L., Yu, N., Chen, E., Liu, H., … Liu, T. Y. (2024). TamGen: drug design with target-aware molecule generation through a chemical language model. Nature communications, 15(1), 9360. https://doi.org/10.1038/s41467-024-53632-4
11 Qi, X., Zhao, L., Tian, C., Li, Y., Chen, Z. L., Huo, P., Chen, R., Liu, X., Wan, B., Yang, S., & Zhao, Y. (2024). Predicting transcriptional responses to novel chemical perturbations using deep generative model for drug discovery. Nature communications, 15(1), 9256. https://doi.org/10.1038/s41467-024-53457-1
12 Liu, X., Wang, Q., Zhou, M., Wang, Y., Wang, X., Zhou, X., & Song, Q. (2024). DrugFormer: Graph-Enhanced Language Model to Predict Drug Sensitivity. Advanced science (Weinheim, Baden-Wurttemberg, Germany), 11(40), e2405861. https://doi.org/10.1002/advs.202405861
13 Kabir, A., Bhattarai, M., Peterson, S., Najman-Licht, Y., Rasmussen, K. Ø., Shehu, A., Bishop, A. R., Alexandrov, B., & Usheva, A. (2024). DNA breathing integration with deep learning foundational model advances genome-wide binding prediction of human transcription factors. Nucleic acids research, 52(19), e91. https://doi.org/10.1093/nar/gkae783
14 Shao, B., & Yan, J. (2024). A long-context language model for deciphering and generating bacteriophage genomes. Nature communications, 15(1), 9392. https://doi.org/10.1038/s41467-024-53759-4
15 Pareja, F., Dopeso, H., Wang, Y. K., Gazzo, A. M., Brown, D. N., Banerjee, M., Selenica, P., Bernhard, J. H., Derakhshan, F., da Silva, E. M., Colon-Cartagena, L., Basili, T., Marra, A., Sue, J., Ye, Q., Da Cruz Paula, A., Yeni Yildirim, S., Pei, X., Safonov, A., Green, H., … Reis-Filho, J. S. (2024). A Genomics-Driven Artificial Intelligence-Based Model Classifies Breast Invasive Lobular Carcinoma and Discovers CDH1 Inactivating Mechanisms. Cancer research, 84(20), 3478–3489. https://doi.org/10.1158/0008-5472.CAN-24-1322
16 Sun, X., Wu, Z., Su, J., & Li, C. (2024). A deep attention model for wide-genome protein-peptide binding affinity prediction at a sequence level. International journal of biological macromolecules, 276(Pt 2), 133811. https://doi.org/10.1016/j.ijbiomac.2024.133811
17 Cai, S., Venugopalan, S., Seaver, K., Xiao, X., Tomanek, K., Jalasutram, S., Morris, M. R., Kane, S., Narayanan, A., MacDonald, R. L., Kornman, E., Vance, D., Casey, B., Gleason, S. M., Nelson, P. Q., & Brenner, M. P. (2024). Using large language models to accelerate communication for eye gaze typing users with ALS. Nature communications, 15(1), 9449. https://doi.org/10.1038/s41467-024-53873-3
18 Wan, P., Huang, Z., Tang, W. et al. Outpatient reception via collaboration between nurses and a large language model: a randomized controlled trial. Nat Med 30, 2878–2885 (2024). https://doi.org/10.1038/s41591-024-03148-7
