基于深度学习,通过病理切片直接预测HPV状态|文献速递·24-09-16
小罗碎碎念
有段时间没有写文献速递的推文了,搞得自己今天写还怪不适应的。
今天所有的推文,都是围绕一个系统的问题展开——既研究了HPV与EBV在头颈癌/鼻咽癌中的致病机制,也总结了如何结合病理组学直接由WSI预测HPV状态——没办法,研究EBV的顶刊就一篇几年前的Nature Communication。
一、利用常规组织切片图像预测头颈癌HPV状态的深度学习技术

一作&通讯
| 角色 | 姓名 | 单位名称(中文) | 单位名称(英文) |
|---|---|---|---|
| 第一作者 | Ruoyu Wang | 英国华威大学计算机科学系,组织图像分析中心 | Department of Computer Science, Tissue Image Analytics Centre, University of Warwick, Coventry, United Kingdom |
| 通讯作者 | Nasir Rajpoot | 英国华威大学生物医学科学学院,沃里克医学院;图灵研究所 | Division of Biomedical Sciences, Warwick Medical School, University of Warwick, Coventry, United Kingdom; the Alan Turing Institute, London, United Kingdom |
文献概述
这篇文章提出了一种基于深度学习的算法,通过分析常规的苏木精-伊红染色组织切片图像,预测头颈癌患者中的人乳头瘤病毒(HPV)感染状态。
研究背景
头颈鳞状细胞癌(HNSCC)的发病机制涉及多种致癌物,如酒精、烟草和HPV感染。HPV感染对HNSCC患者的预后、治疗和生存有显著影响,因此确定肿瘤的HPV状态非常重要。
研究方法
研究者提出了一个深度学习流程,使用ResNet-18作为特征提取器,并通过弱标签策略训练模型。
然后,研究者提出了一个基于三元组排序的训练流程,使用多实例学习(MIL)分类器对每个图像块进行评分,并将评分聚合为全玻片级别的Digital-HPV评分。
此外,研究者还对肿瘤微环境(TME)进行了定量分析,以了解HPV阳性和HPV阴性患者之间的不同免疫反应模式。
研究结果
研究显示,Digital-HPV评分在HPV检测中表现出了最先进的性能,并且与分子检测的HPV状态相比,Digital-HPV评分在生存分析中提供了可比的患者分层。
此外,通过免疫分析和TME的空间分析,研究者发现HPV阳性患者中淋巴细胞浸润水平较高,B细胞和T细胞水平较高,而巨噬细胞水平较低。
结论
这项研究表明,所提出的深度学习算法能够有效地预测头颈癌患者中的HPV感染状态,可能有助于快速且低成本地对患者进行分层,从而优化治疗策略。研究还强调了对肿瘤微环境的详细定量分析的重要性,这有助于开发新的治疗方法和生物标志物。
文章还提到了一些技术细节,如使用的数据集、数据预处理、深度学习模型的具体结构和训练方法、以及实验设置和统计分析方法。此外,文章还讨论了研究的潜在临床应用和未来研究方向。
重点关注
图1展示了文章中提出的深度学习流程,用于预测头颈癌组织切片中的人乳头瘤病毒(HPV)感染状态。
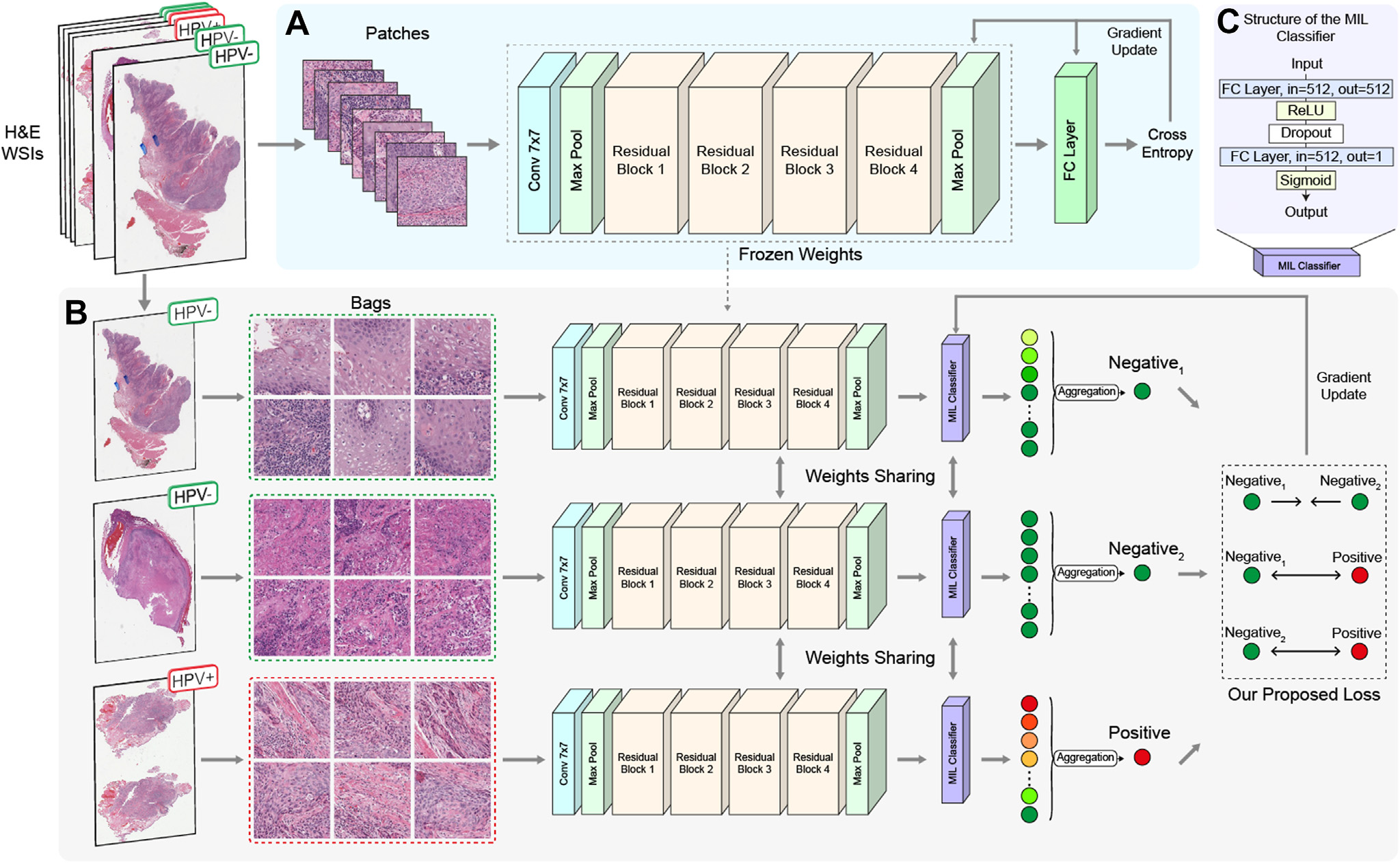
这个流程包含三个主要部分:
A. 特征提取器的训练(弱标签策略):
- 从全玻片图像(WSIs)中提取图像块(patches),这些图像块带有整个玻片的标签。
- 使用预训练的ResNet-18模型来训练一个图像块分类模型,这个模型能够从图像块中提取特征。
B. 三元组排序训练流程:
- 同一WSI中提取的图像块被分为一组(一个“袋子”),并赋予该WSI的标签。
- 使用多实例学习(MIL)分类器对每个图像块进行评分。
- 通过聚合图像块级别的评分来生成全玻片级别的Digital-HPV评分。
- 使用新提出的三元组排序损失函数来计算损失,以便在训练过程中优化模型。
C. MIL分类器的详细结构:
- 该结构包括卷积层(Conv)用于特征提取,全连接层(FC)用于分类,以及ReLU(修正线性单元)作为激活函数。
- 使用最大池化(Max)操作来整合特征。
- 整个结构旨在从苏木精-伊红(H&E)染色的组织切片图像中识别与HPV感染相关的特征。
总的来说,图1描述了一个利用深度学习对病理切片图像进行分析,以预测HPV感染状态的完整流程。这个流程结合了图像块级别的特征学习和全玻片级别的评分聚合,通过三元组排序损失函数来优化模型的预测性能。
二、利用深度学习技术识别头颈部鳞状细胞癌中HPV阳性患者

一作&通讯
| 角色 | 姓名 | 单位 | 单位(中文) |
|---|---|---|---|
| 第一作者 | Sebastian Klein | Institute of Pathology, Medical Faculty, University Hospital Cologne, Cologne, Germany | 德国科隆大学医学院病理研究所,科隆大学医院 |
| 通讯作者 | Sebastian Klein | Gerhard-Domagk-Institute of Pathology, University Hospital Münster, Münster, Germany | 德国明斯特大学医学院格哈德-多马克病理研究所,明斯特大学医院 |
文献概述
这篇文章是关于一项研究,该研究利用深度学习技术来预测头颈部鳞状细胞癌(OPSCC)患者组织样本中人类乳头瘤病毒(HPV)的存在,并识别出预后良好的患者。
研究的主要内容包括:
-
目的:HPV与OPSCC的发生有关,并且与较好的预后相关。研究旨在开发一种基于深度学习的方法,通过常规的苏木精-伊红(H&E)染色切片来预测HPV的关联性。
-
实验设计:研究者生成了一个基于深度学习的HPV预测评分(HPV-ps),并评估了其在预测HPV关联性方面的性能。研究使用了来自两个不同地点的273名患者的数据,然后评估了在总共594名患者中的预后相关性。
-
结果:尽管病理学家能够通过H&E染色切片诊断HPV关联(平均AUC为0.74),但与其他三位观察者相比,一致性较低(Light Kappa为0.37)。相比之下,HPV-ps在两个独立队列中的平均AUC为0.8。HPV-ps在三个不同队列的594名患者中识别出了预后良好的个体。
-
结论:使用深度学习技术结合常规H&E染色切片来检测OPSCC中的HPV关联,可以作为一种单一的生物标志物,或与p16状态结合使用,以识别预后良好的OPSCC患者,可能比结合HPV-DNA/p16状态作为患者分层的生物标志物更为有效。
-
研究方法:包括患者选择、HPV-DNA和p16状态的评估、全切片图像的处理和统计分析等。
-
专家评审过程:四名经验丰富的病理学家对152例OPSCC进行了HPV状态的评估,并通过虚拟全切片图像进行分类。
-
训练数据集:为了提高模型的泛化能力,训练数据集包括了TCGA数据和两个不同地点的全切片图像。
-
研究意义:研究表明,通过深度学习算法预测的HPV状态可以作为识别预后良好患者的有效工具,并且可能对临床治疗决策有重要影响。
文章还讨论了研究的意义、作者贡献、资金支持以及利益冲突声明。
重点关注
图1提供了这项研究中使用的算法和研究方法的概览,具体可以分为两个主要部分:
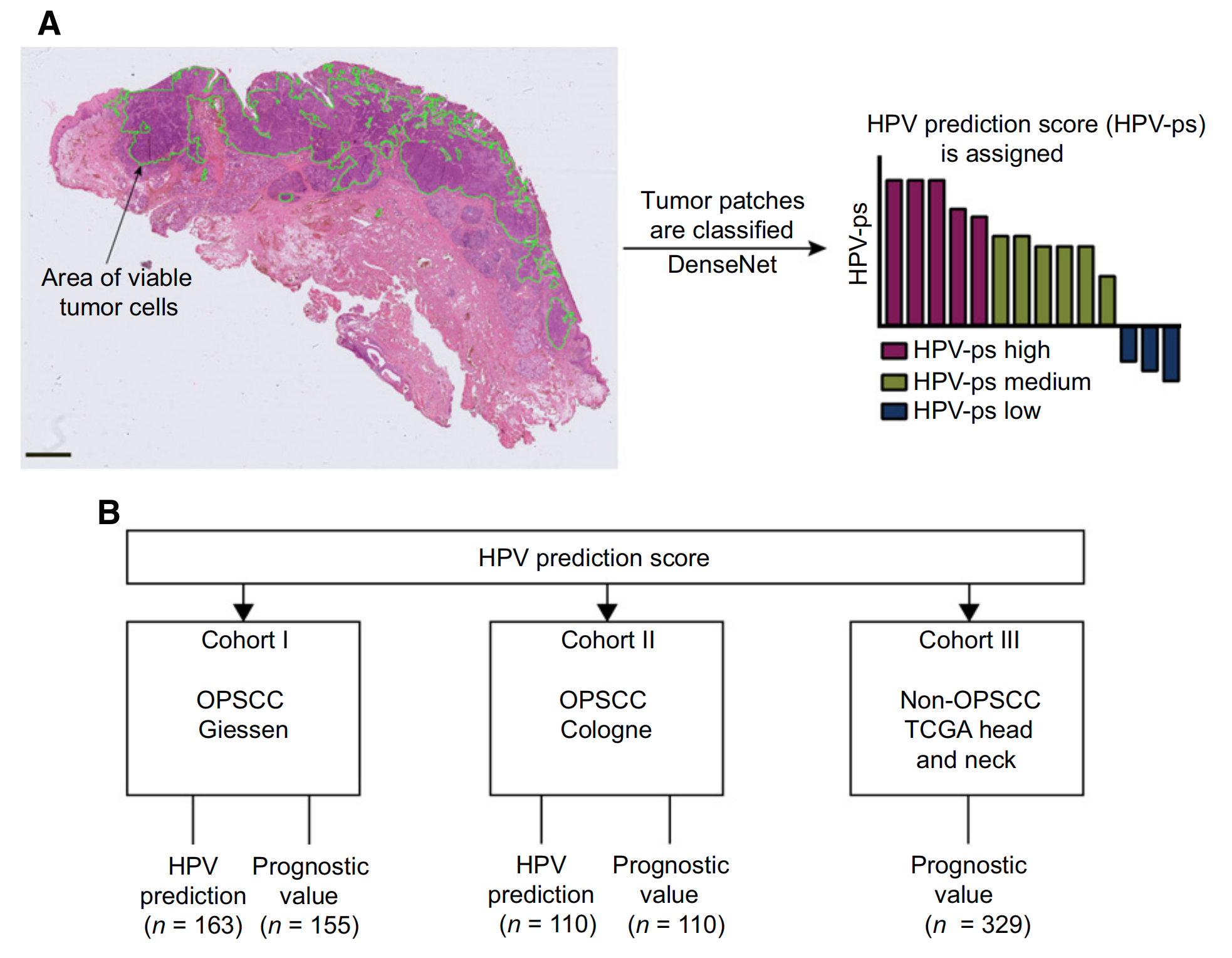
A部分:算法流程
- U-Net深度卷积神经网络:首先,使用U-Net这种深度学习架构来检测OPSCC(口咽鳞状细胞癌)和HNSCC(头颈部鳞状细胞癌)中的活性肿瘤区域。U-Net是一种常用于图像分割的网络,它能够识别并分割出图像中的特定区域。
- 肿瘤区域提取:通过U-Net识别出的活性肿瘤区域(图中用绿色线条圈出)被提取出来,形成图像块(称为肿瘤块或肿瘤补丁)。
- DenseNet分类:提取出的肿瘤块随后被输入到DenseNet网络中进行分类。DenseNet是一种密集连接的卷积神经网络,它能够基于学习到的特征对肿瘤块进行分类,并给出每个肿瘤块的HPV预测评分(HPV-ps)。
- HPV预测评分(HPV-ps):DenseNet网络为每个肿瘤块分配一个评分,这个评分反映了该肿瘤块与HPV关联的可能性。评分越高,表示肿瘤块与HPV关联的可能性越大。
B部分:研究方法概览
- 三个队列分析:研究中分析了三个不同的患者队列,总共包括594名患者。这些队列可能来自不同的地理位置或研究组,旨在验证算法的泛化能力和预测准确性。
- 队列分析的目的:通过在不同的患者群体中应用和测试HPV-ps,研究者能够评估该评分在实际临床环境中的应用潜力和预后价值。
整体而言,图1展示了一个结合了图像处理和机器学习的多步骤流程,用于从常规的病理切片中自动检测和预测HPV在头颈部癌症中的关联性,进而可能影响临床治疗决策和患者预后评估。
三、低成本数字病理学与深度学习模型的结合

一作&通讯
| 角色 | 姓名 | 单位名称(英文) | 单位名称(中文) |
|---|---|---|---|
| 第一作者 | Divya Choudhury | Pritzker School of Medicine, University of Chicago | 芝加哥大学普里茨克医学院 |
| 通讯作者 | Alexander T. Pearson | Section of Hematology/Oncology, The University of Chicago Medicine & Biological Sciences | 芝加哥大学医学中心血液学/肿瘤学部 |
文献概述
这篇文章介绍了一种低成本、开源的工作站和计算流程,利用深度学习技术自动进行组织病理学评估,以提高资源有限地区癌症诊断的可及性和准确性。
研究团队由来自不同机构的成员组成,包括芝加哥大学、Geisinger癌症研究所、芝加哥大学医学中心、德保罗大学、威斯康星大学麦迪逊分校以及德国的几所研究机构。
背景
在资源有限的环境中,部署和获取最先进的精准医疗技术仍然是提供公平的全球癌症护理的一个基本挑战。随着数字病理学的发展,它与诊断人工智能算法的潜在结合提供了一个机会,可以民主化获取个性化医疗的机会。然而,目前的数字病理工作站成本高昂。随着许多中低收入国家癌症发病率的上升,验证和实施低成本自动化诊断工具对于帮助医疗保健提供者管理日益增长的癌症负担至关重要。
方法
研究团队描述了一个低成本(230美元)的工作站,用于数字幻灯片捕获和计算分析,由开源组件组成。他们分析了在使用这种开源工作站捕获的病理图像上使用深度学习模型与使用更昂贵的硬件捕获的图像上的性能。
发现
与传统的病理图像捕获方法相比,使用开源工作站进行的低成本数字幻灯片捕获和分析(包括低成本显微镜设备)与模型性能在乳腺癌、肺癌和头颈癌分类上具有可比的准确性。在患者水平分析中,头颈癌HPV状态预测的AUROC为0.84,肺癌亚型预测为1.0,乳腺癌分类为0.80。
研究内容
文章还讨论了全球癌症负担的增加,以及如何通过适应高资源环境中使用的先进癌症诊断工具来解决癌症护理中的不平等问题。数字病理学的优势包括远程协作、大量可分析数据的轻松获取、快速诊断、减少收集和存储物理玻片的负担以及减少人类病理学家审查所需的成本。深度学习算法有潜力自动化诊断工作流程,减少成本,同时提供与人类病理学家在组织图像中识别的相同信息。
研究背景
研究团队通过搜索PubMed和Google Scholar,找到了涉及低成本显微镜和/或数字病理学、机器学习或深度学习模块的文献。他们指出,尽管已有文献讨论了基于深度学习的诊断和临床工具的开发,但很少有论文讨论在低成本和开源硬件和软件上验证现有深度学习模型用于组织病理学癌症亚型的研究。
研究价值
这项工作的目的是展示如何将现有的深度学习方法与完全低成本、开源的工具结合起来,同时保持模型的准确性。他们的发现作为概念验证,表明可以使用深度学习模型对低分辨率的病理图像进行分类,并且可以在低成本的计算硬件上成功运行高性能的深度学习模型。
研究结果
文章详细描述了如何使用开源工具和低成本硬件来构建一个完整的数字病理学深度学习分析流程。研究团队开发并测试了一个230美元的平台,用于捕获组织病理学幻灯片的图像,并使用在55美元的Raspberry Pi计算机上运行的开源深度学习管道对来自三个不同数据集的组织样本进行分类。
方法
数据收集用于训练深度学习模型执行一组不同的病理分类任务。研究团队选择了与已发布或预印数据相匹配的深度学习模型,包括TCGA中头颈癌的HPV状态、肺癌和乳腺癌亚型。
他们收集了公开的组织学图像,这些图像使用Aperio ScanScope或其他Aperio幻灯片扫描仪捕获并存储在SVS格式中。为了在模型训练中使用回顾性数据,他们从TCGA收集了472个已知HPV状态的头颈癌患者的数字化WSI组织样本。
对于模型测试,他们使用了来自UCMC的外部验证数据集,包括来自已知HPV状态的头颈癌患者的十个组织病理学玻片。
图像处理
为了评估低成本与高成本图像捕获方法的影响,他们为UCMC验证队列制作了两组扫描的组织病理学图像:1)临床级显微镜的WSI,以及2)低成本显微镜的部分幻灯片图像。他们使用Slideflow软件对图像进行了预处理,包括Reinhard-Fast染色标准化。
深度学习模型
他们训练了深度学习分类模型,用于TCGA头颈癌病例中HPV阳性与阴性状态的分类,TCGA肺癌病例中的肺腺癌与鳞癌,以及TCGA乳腺癌队列中的乳腺小叶癌与导管癌。然后使用来自UCMC的验证集(每个类别五个幻灯片)来验证每个模型。
结果
在Raspberry Pi上,他们对24种模型架构、四种不同的瓦片尺寸和多种批量大小进行了深度学习推理速度的基准测试。使用Xception架构(在299×299像素)的可识别模型允许以每秒1.04张图像的速度进行预测。所有在10×放大倍率下使用低成本显微镜捕获的原始图像都表现出一定程度的模糊、颜色失真和/或球面像差。使用整个幻灯片用户界面,可以以大约每秒1张图像的速度生成模型训练在299×299像素图像上的焦点预测。
讨论
开源技术确保了更广泛的提供者和社区能够获得基于人工智能的诊断工具。研究结果证明了在大幅降低图像采集和计算硬件成本的同时保持模型准确性的可行性。他们展示了使用低成本开源硬件进行图像采集和计算步骤的可行性,这些步骤需要将机器学习方法应用于数字病理学和癌症诊断。尽管低成本显微镜的成本比目前使用的临床级显微镜低几个数量级,但模型性能得以维持。
重点关注
图1展示了一个低成本的开源数字病理工作流程,具体内容如下:
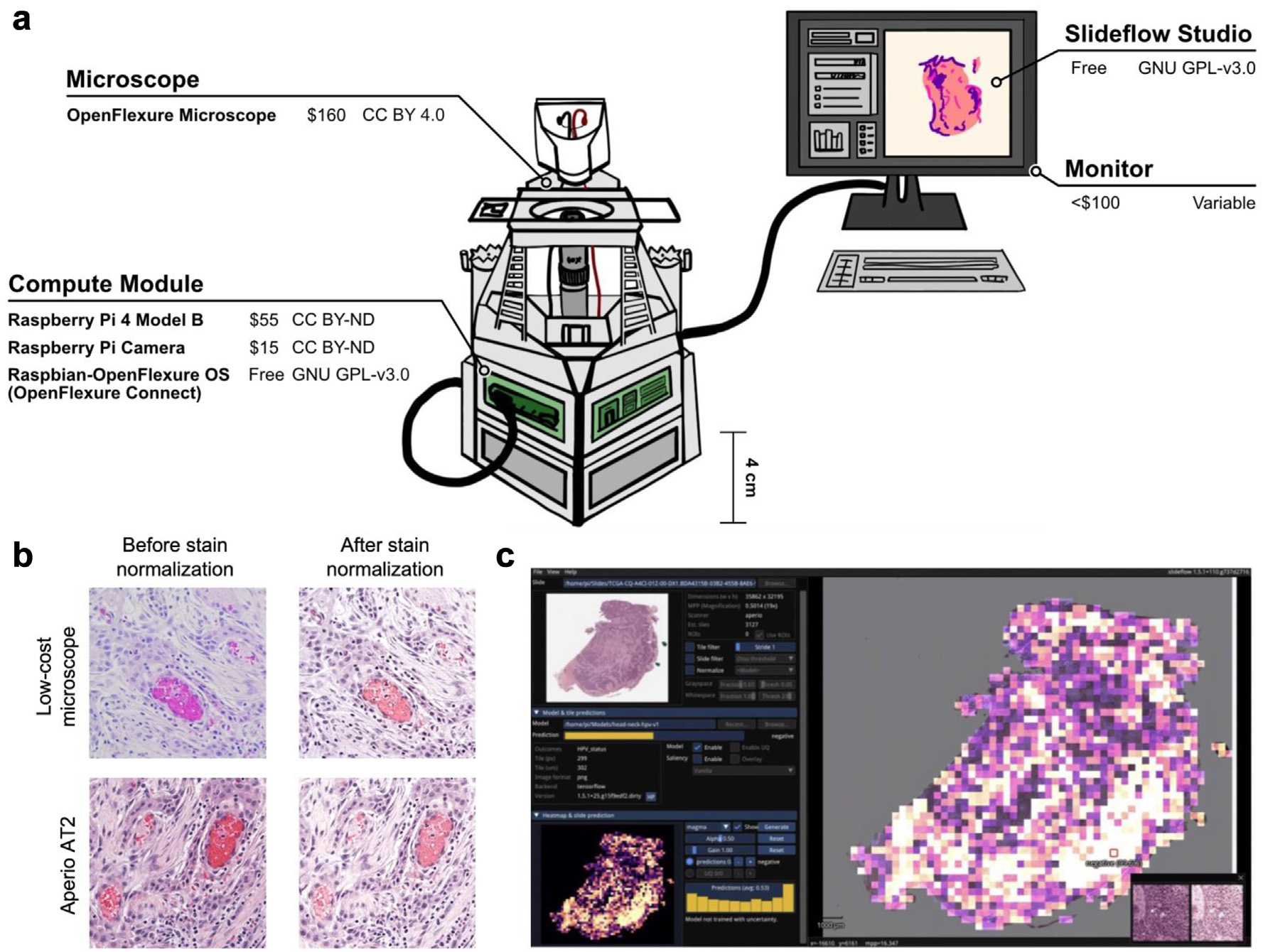
(a) 低成本开源数字病理工作站的组成包括硬件和软件两部分:
- 硬件组件:OpenFlexure显微镜、Raspberry Pi 4 Model B(树莓派4B型号)、Raspberry Pi相机模块和显示器。
- 软件组件:OpenFlexure Connect、Raspberry Pi操作系统和Slideflow。
- 这些组件的成本和许可证信息在图中给出。
(b) 开源用户界面用于交互式可视化和生成模型预测:
- Slideflow软件能够部署多种训练好的模型,用于数字病理图像分类。
- 可以为部分幻灯片和整个幻灯片图像生成预测。
- 预测结果可以以热图的形式呈现整个幻灯片,或者以单独的瓦片形式呈现特定区域(如图右下角所示)。
- Slideflow用户界面针对x86和低功耗ARM架构设备进行了优化。
- 上述截图显示了在Raspberry Pi 4B上捕获的整个幻灯片图像(WSI)预测的热图。
© 计算染色标准化的效果:
- 通过计算染色标准化,提高了由Aperio AT2幻灯片扫描仪和低成本OpenFlexure设备捕获的图像之间的视觉相似性。
总体而言,图1强调了低成本开源工作站的可行性,该工作站能够利用开源软件和低成本硬件实现与高成本设备相媲美的数字病理图像分析和预测。这种工作站的建立为资源有限的地区提供了一种经济高效的解决方案,以实现精准的病理诊断。
四、结合细胞形态学和空间邻近性的新型细胞图:在肺癌和HPV相关口咽癌中的应用

一作&通讯
| 作者角色 | 作者姓名 | 单位(英文) | 单位(中文) |
|---|---|---|---|
| 第一作者 | Cheng Lu | Department of Biomedical Engineering, Case Western Reserve University | 凯斯西储大学生物医学工程系 |
| 通讯作者 | Anant Madabhushi | Department of Biomedical Engineering, Case Western Reserve University | 凯斯西储大学生物医学工程系 |
文献概述
这篇文章介绍了一种新的特征驱动的局部细胞图(Feature-driven local cell graph,简称FLocK)方法,用于分析组织病理学图像中的细胞核空间排列,以预测肺癌和口咽癌的临床结果。
FLocK用于分析不同癌症亚型组织病理学图像中细胞核的局部空间排列,这种方法结合了细胞核的空间邻近性和个体属性(如形状、大小、纹理)。研究者设计了一组新的定量图衍生指标,用于从FLocK中提取,以捕捉不同邻近核团簇之间的相互作用。
研究者评估了从苏木精-伊红(H&E)染色组织图像中提取的FLocK特征在两个临床应用中的有效性:一是对早期非小细胞肺癌(ES-NSCLC)患者进行短期与长期生存分类,二是预测口咽鳞状细胞癌(OP-SCC)的人类乳头瘤病毒(HPV)状态。
在对ES-NSCLC患者进行长期与短期生存分类的研究中,通过最小冗余最大相关性(MRMR)选择,确定了与FLocK大小变化和交叉FLocK距离变化相关的前10个区分特征。结合线性判别分类器,在训练队列中得到了预测生存的平均AUC(Area Under Curve,曲线下面积)为0.68,优于其他现有的组织形态计量和深度学习方法。
在对OP-SCC的HPV状态分类中,使用与交叉FLocK部分相关的前三个FLocK特征构建的分类器,在训练队列中得到了0.80的AUC,在独立测试队列中得到了0.78的准确率。将FLocK测量与细胞团簇图、核方向和核形状结合使用,分别将训练AUC提高到了0.87、0.91和0.85。深度学习方法在这一应用中的性能略优于基于FLocK的分类器,但在测试队列中,FLocK的性能与深度学习方法相当。
文章还讨论了FLocK在分析实体肿瘤组织图像方面的独特性和定量方法,以及如何从与现有组织形态计量不同的方面来审视肿瘤形态。研究者提供了FLocK的源代码,以供进一步的研究和应用。
重点关注
图2展示了特征驱动的局部细胞图(FLocK)构建和相关特征计算的工作流程。
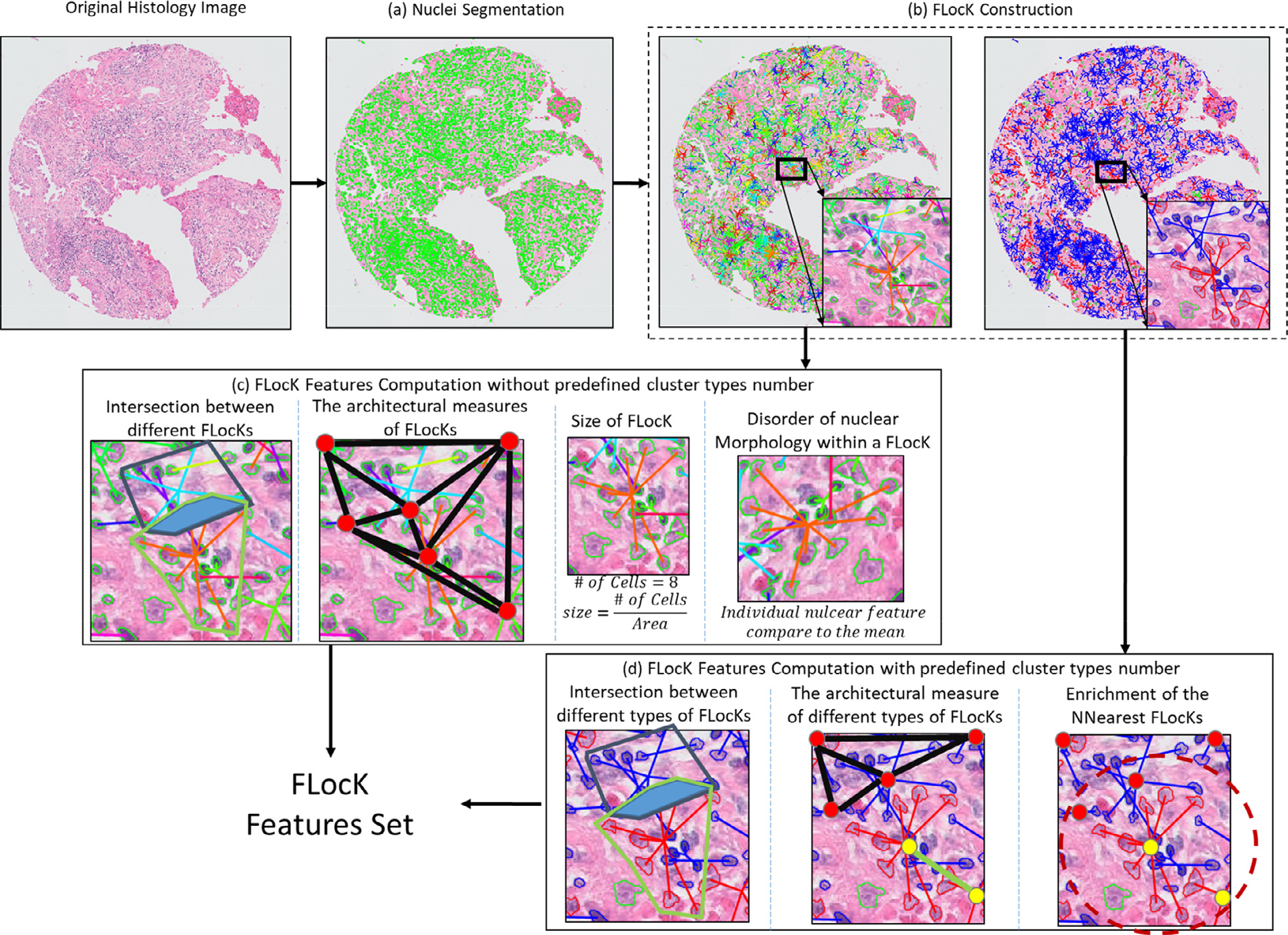
(a) 核边界的识别:在这个阶段,使用图像处理技术来识别和标记图像中的细胞核边界。图中以绿色轮廓表示这些边界,这是后续FLocK构建的基础。
(b) 基于核特征的FLocK构建:在这个阶段,根据细胞核的特征(例如,本例中使用的平均光密度)来构建FLocK。具有相同特征的细胞核被连接起来,形成局部细胞团簇,并且属于同一FLocK的细胞核之间有相同颜色的连接边。这种构建方式允许研究者探究具有相似形态学特征的细胞核之间的局部空间关系。
© 无预定义聚类类型的FLocK特征计算:在没有预先设定聚类类型数量的情况下,通过分析构建的FLocK来计算特征。这可能包括计算FLocK的大小、形状、内部细胞核的排列方式等特征,以及FLocK之间的空间关系,如交叉和邻近关系。
(d) 有预定义聚类类型的FLocK特征计算:在已知聚类类型数量的情况下进行FLocK特征的计算。这种方法允许研究者根据特定的生物学假设或先验知识,将细胞核分组到特定的FLocK中,并计算与这些预定义聚类相关的特征,如不同类型FLocK之间的交互作用。
总的来说,图2描述了从图像中识别细胞核边界、基于核特征构建FLocK、以及计算FLocK特征的整个过程。这些特征计算可以为病理学图像分析提供定量的、可能与疾病预后相关的信息。通过比较有无预定义聚类类型的方法,研究者可以探索不同构建策略对特征提取和临床应用的影响。
