使用多模态数据映射大脑网络
前言
人脑由解剖和功能性的网络组织而成,这些网络涉及大规模分布但相互作用的脑区。不同脑区之间的同步性已在手指敲击或视觉刺激等实验任务活动中观察到,但更重要的是,也在无任务条件下(即静息态)测量的内源性活动中被观察到。即使在休息时,当人们清醒但没有有意识地执行任何脑力或体力任务时,人脑也处于活跃状态,并伴随有时空组织的神经活动波动。因此,在静息态人脑中观察到的这些网络组织被称为静息态网络(RSNs),内在连接网络(ICNs)或内在大脑网络。本文主要使用术语ICNs而不是RSNs,是因为这些网络组织表明了人脑的内在属性。此外,在其他物种中也发现了类似的内在大脑网络,例如大鼠和非人类灵长类动物,这表明这些大脑网络在哺乳动物物种中的普遍存在,以及它们在理解人类大脑如何工作以及大脑如何支持人类行为方面是非常重要的。
在许多基础神经科学研究中,已经发现内在的大脑网络不是孤立的大脑区域,而是各种神经过程以及随后的行为或精神状态的分布式神经基质。据广泛报道,几乎所有神经系统和精神疾病均存在ICN异常,特别是重度抑郁症、阿尔茨海默症、精神分裂症、自闭症和癫痫。这些研究表明了ICN在神经精神疾病中的诊断价值。更重要的是,最近的许多研究进一步证明,神经精神障碍患者改变的大脑网络可以通过神经刺激技术(例如经颅磁刺激(TMS))缓解症状或正常化。这些研究表明,靶向内在的大脑网络是治疗神经精神疾病的一种有效手段,可替代药物进行治疗。因此,内在大脑网络在基础神经科学和临床神经科学中都具有重要的意义。
人脑网络成像可采用各种功能性神经成像方式实现,包括功能性磁共振成像(fMRI),电生理记录,如脑电图(EEG),皮质电图(ECoG)和脑磁图(MEG),以及正电子发射断层扫描(PET)和功能近红外光谱(fNIRS)。其中,fMRI因其空间穿透力和优越的空间分辨率而在脑网映射中起着主导作用。然而,由于血流动力学响应的缓慢特性,其时间分辨率较低。EEG和MEG是两种用于映射大脑功能和网络的非侵入性表面电生理记录技术,它们能够提供实时的时间信息,但空间穿透力和分辨率有限。PET被用于评估大脑网络的历史悠久,甚至早于fMRI技术,但它使用了潜在有害的放射性物质,并且时间分辨率比fMRI更差。与fMRI一样,fNIRS测量的是血流动力学信号,但由于其采样频率更快,因此具有比fMRI更好的时间分辨率。然而,由于fNIRS是一种像EEG/MEG一样的表面记录技术,因而其空间分辨率比fMRI低。从设备的便携性角度来看,EEG和fNIRS是两种允许在自然环境中进行长时间记录的模式,尽管便携式MEG正在开发中。对于每种模式,已经提出了许多分析方法来量化不同大脑区域之间的功能连接网络,例如,数据驱动分析和基于种子的分析。
除了用于映射大脑网络的单一成像模式之外,同步记录的多模态数据也被用于研究大脑网络,以获得“整体大于部分之和”的知识信息,因为每种模式对人脑如何工作的特定方面都很敏感,将它们结合起来可以解决每种模式的局限性,并提供关于人脑网络更全面的图像。例如,电和血流动力学信号或者代谢和血流动力学信号可分别通过同步EEG和fMRI或者PET和MRI记录相组合。此外,电、血流动力学和代谢信息可以通过同步三模态EEG、fMRI和PET记录来整合。
使用fMRI的脑网络映射
fMRI作为一种成熟的模式,是脑网络功能成像的主要工具,因为它具有毫米级精度的高空间分辨率,并且能够可靠地检测大脑深部区域的神经活动变化。fMRI利用血氧水平依赖(BOLD)对比成像,对生理变化(包括脑血流、脑血容量和脑氧代谢等)的复杂相互作用非常敏感。在BOLD fMRI首次进行功能映射后不久,在无任务的静息态下对人脑进行成像,揭示了大幅和低频振荡(<0.1Hz)形式的活动。更重要的是,这种自发活动在时间上与远处的大脑区域相关,例如,休息时横跨左右半球的运动网络。自此,基于内源性静息态信号的不同脑区之间的时间相关性被称为静息态功能连接(RSFC)。具有显著高RSFC值的脑区也被统称为一个网络,即RSNs或ICNs。
由于fMRI信号是4D数据,包括3D空间体积和1D时间,因此选择合适的信号处理方法是表征ICNs的关键。已经提出了许多数据分析方法来识别fMRI信号中的ICNs,这些方法大致可分为两类:假设驱动法和数据驱动法(也分别称为模型依赖法和无模型法)。前一种方法包括基于种子的方法,该方法检查种子脑区相对于所有其他脑区的功能连接,并生成关于预定义种子区域的功能连接图。在这里,可以根据解剖信息选择种子区域,也可以根据用于测试特定假设的任务实验中的大脑激活来选择种子区域。例如,可以选择手指敲击任务中激活最大的运动区域上的体素作为种子来研究静息态下的运动网络。为了量化基于种子的方法中的功能连接,通常会使用相关性分析。在获得功能连接图后,通常使用具有任意值或通过统计分析获得的阈值来识别与种子显著相关的体素。基于种子的方法简单明了,结果也相对容易解释。但是,由于该方法需要对种子进行先验选择,因此结果可能会因所选种子的质量而有所偏差。此外,由于种子是单独选择的,这种数据分析方法无法在一次分析中揭示全脑尺度上的多种功能连接模式。
为了解决假设驱动法的局限性,目前已提出了一些数据驱动的方法,包括独立成分分析(ICA)和聚类方法。这些方法完全由fMRI数据驱动,无需进行先验假设。其中,最流行的方法是ICA,这是一种盲源分离技术,可最大限度地提高分解成分信号之间的统计独立性。对于fMRI数据,通常对4D数据进行空间ICA,以识别一组空间独立的成分。每个成分都有其空间图和时间波形图,用于判断该成分是否代表网络的大脑活动或者由于测量或生理噪声而产生的伪影。因此,与基于种子的方法相比,需要额外的步骤来识别与实际大脑活动相对应的成分。尽管如此,ICA是探索fMRI数据中大脑网络最常用的方法,因为它能够在一次分析中从全脑数据中识别多个ICNs,这与基于种子的方法不同。第二个众所周知的数据驱动法是聚类方法。聚类方法根据Pearson相关、L2距离和L1距离等空间域相似性指标,沿时间轴将全脑体素数据的不同帧分类为不同的子组。获得子组后,根据分类结果定义每个子组的二进制时间进程。然后,通过扫描所有体素,并使用单个体素时间进程和二进制时间进程之间的额外相关分析步骤来识别每个子组的大脑网络的空间模式。此外,为了充分利用两类方法的优点,目前提出了一些混合方法,例如混合基于ICA-种子的方法。
尽管在不同的情况下使用不同的方法来表征大脑网络,但在许多使用不同方法的fMRI研究中报告的ICNs的空间模式对于健康被试相应的大脑网络是非常一致的。目前,一些关键的内在脑网络已经在文献中得到了广泛的认可(见图1),包括默认模式网络(DMN),感觉运动网络,视觉网络,听觉网络和额顶叶网络。每个网络都涉及解剖学上分离但功能上相互连接的脑区,这些区域在BOLD信号中显示出高度的相关性。更重要的是,这些在无任务(即静息态)条件下获得的网络与通过任务或刺激范例建立的已知功能网络高度相似。例如,感觉运动和视觉网络分别与初级运动皮层和初级视觉皮层具有高度重叠的空间映射。其中,DMN的特征与大多数其他ICN明显不同。虽然大多数ICN涉及一些功能特定的脑区,但DMN由多个功能相连但距离较远的脑区组成(图1中的映射图420),这些区域包括后扣带皮层/楔前叶、内侧前额叶皮层和顶下叶。
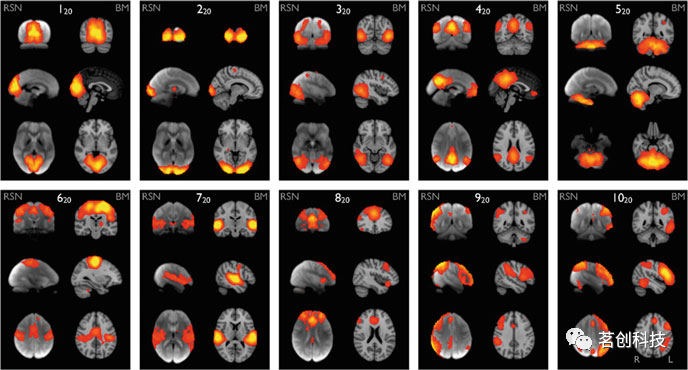
图1.从29671名被试的BrainMap(BM)激活数据库(每对右列)和36名被试的静息态fMRI数据集(每对左列)的ICA中确定了10个具有代表性的RSNs。
虽然fMRI的脑网络最初被提出作为一种研究健康人脑功能结构的手段,但目前已进行了大量研究以探索其在各种神经和精神疾病中的临床应用。阿尔茨海默症(AD)、精神分裂症、自闭症谱系障碍(ASDs)、抑郁症、意识障碍、创伤后应激障碍(PTSD)和平衡障碍等几乎所有脑部疾病都研究了多个大脑网络的性质。从所有这些研究中,可以发现DMN在神经和精神疾病中具有特殊的意义,因为DMN与人类认知的核心过程有关。在不同的脑部疾病中,已确定的脑网络异常在研究和人群中报告了不同程度的一致性。阿尔茨海默症是最早被认为具有重要临床价值的脑部疾病之一,因为正常衰老和AD都显示出人脑结构和功能的深刻变化。这些研究大多发现AD相关痴呆患者的DMN、突显和边缘脑网络的一致改变,其中大多数一致的异常被认为与DMN有关(参见图2中AD组与健康老年组相比,AD组的DMN活性降低)。研究表明,上述异常内在脑网络中高度连接的中枢区域可能是AD的早期损伤靶点。相比之下,精神分裂症,另一种引起大量fMRI脑网络研究的脑部疾病,在其脑网络模式异常发现方面存在争议。精神分裂症患者的听觉、DMN、感觉运动网络均存在明显的低连接。然而,也有研究称DMN的功能连接增加,这与精神分裂症的主要症状(即思维障碍)一致。综上所述,使用静息态脑网络的临床研究正在快速增长,其目标是确定特定脑网络作为疾病评估和治疗监测的生物标志物的诊断和预后价值。
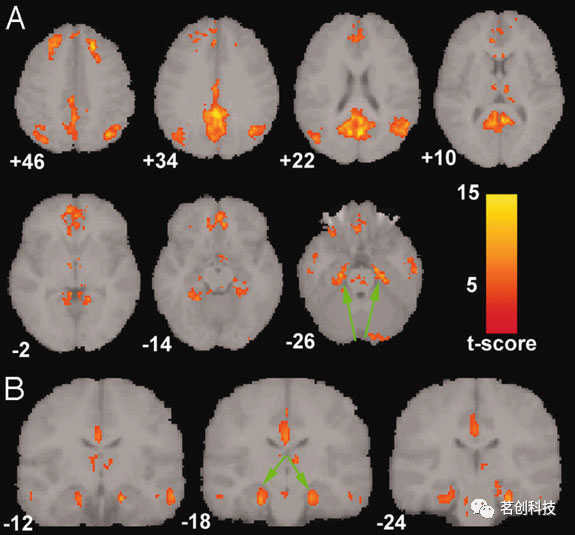
图2.与AD老年人相比,健康老年人的DMN活性增加。
使用EEG和MEG的脑网络映射
与使用fMRI的ICN研究相比,使用电生理信号映射内在大脑网络的综合研究发展相对较晚。电生理信号的优势在于它们直接测量神经元电活动,而不是继发性血流动力学,电生理信号具有毫秒级的高时间分辨率。在不同的电生理信号中,EEG和MEG是测量人脑信号的两种非侵入性方式。使用EEG和MEG信号,有关大脑网络的信息首次直接在表面传感器领域的记录数据中进行了探索,与下面将讨论的源域形成对比。EEG微状态识别,即静息态EEG信号中反复出现的空间地形模式,通常持续约80-100ms,是表征EEG传感域与脑网络相关的空间模式的一种手段。虽然对这些EEG微状态的探索没有考虑到脑网络的概念,但有一些研究已经将EEG微状态与fMRI研究确定的ICNs联系起来。例如,Britz等人(2010)发现四种EEG微状态,即A、B、C、D,分别与语音处理、视觉网络、突显网络和注意网络的ICN相关。Yuan等人(2012)从EEG中识别出13个微状态,从fMRI中识别出10个ICNs,并通过计算ICNs的时域动态相关性发现每个微状态都与一些ICNs相关联。此外,ICA也被用于表征EEG ICNs,在传感域中识别的单个IC被用于表示潜在同步神经元活动产生的信号。然而,在传感域EEG信号中报告的大脑网络并不能提供有关其潜在神经元活动或大脑活动网络模式的直接信息,因为这些已识别的与大脑网络相关的传感器水平模式只是潜在分布式神经源的表面替代物。因此,在传感器水平上比较EEG信号和fMRI信号之间的脑网络是非常重要的。此外,EEG ICs与偶极源有关,这为ICs的网络解释提出了另一种(但相互矛盾的)理论。除了这些问题和争论之外,还有两个与用于研究功能连接的传感器信号相关的方法问题:①“场扩散”问题,即多个传感器接收来自同一源的信号;②由于电传导引起的“模糊”效应。这两个问题都可能导致对实际功能连接的错误估计。有研究发现,在传感域表征的脑网络和在源域表征的脑网络在其网络拓扑结构上的相关性较弱。因此,有越来越多的研究使用EEG和MEG信号探索源域中的大脑网络。
图3描述了在EEG/MEG源域中表征大脑网络的一般过程。首先,需要从传感器级的EEG或MEG数据重建脑源数据,通过求解所谓的逆问题来解决场扩散问题和模糊效应(图3A)。为了准确估计源数据,需要考虑四个方面:①高密度记录的EEG或MEG信号,例如至少32个通道,并优先选择超过64个通道的数据;②根据显示个体大脑结构的MRI数据量身定制并准确建立头部体积导体模型和皮层源模型;③传感器/通道位置需要数字化,并与基于MRI数据构建的模型精确地共配准;④解决逆问题的好方法。最后一个点是最重要的组成部分,主要决定源估计的性能,因为从数学上讲,逆问题是一个没有唯一解的不适定问题。为了解决逆问题的多解性和不适定性,已有文献提出了许多方法,这些方法基于所需源特征或生理约束的先验信息,如最小范数法、波束形成法和稀疏源成像法。其次,需要使用适当的模式识别方法从重建的源数据中识别大脑网络(图3B),该任务类似于在fMRI数据中识别ICNs。由于皮层表面或脑容积上的电生理源数据的表征与fMRI数据相当,因此可以类似地应用fMRI中推导脑网络的方法。事实上,大脑网络可以直接在电生理源数据上进行表征。但由于所谓的信号泄漏问题,可能会导致不同脑区之间的重影相互作用。在量化不同脑区之间的通信时,提出了三种方法来解决这一问题。第一种是使用连接测量,排除了脑区之间瞬时、零滞后相互作用的影响。第二种是在表征脑网络之前对重建的源数据执行额外的处理步骤,目的是消除零滞后的相互作用,例如所谓的“正交化”。然而,上述两种方法的局限性在于,零滞后的相互作用可能不仅仅是由容积传导效应引起的,而是实际的神经元相互作用。第三种方法是在计算连接测量后使用额外的统计方法(图3C),以便通过统计手段识别大脑网络。当然,这些统计方法需要精心设计,以表示非假设条件下连接测量分布中的容积传导效应。
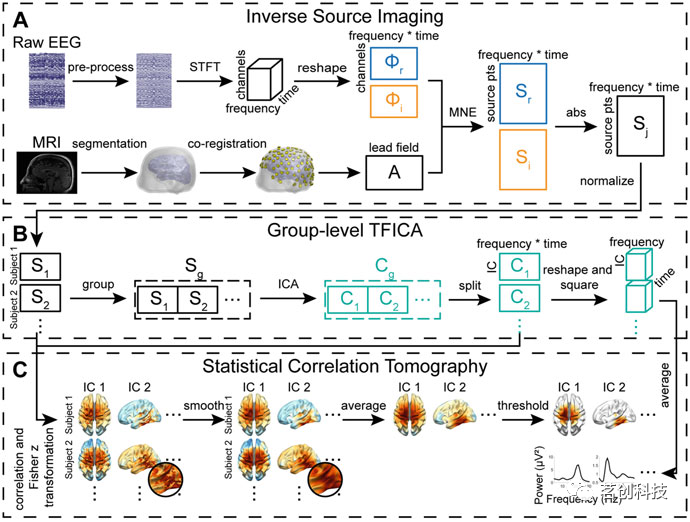
图3.重建源域ICNs的时频ICA(TFICA)-统计相关层析成像(SCT)示意图。
同样,fMRI数据的数据驱动方法也可以应用于EEG/MEG源数据,以提取大脑网络模式。例如,ICA已被广泛用于识别EEG/MEG脑网络的源数据。与假设驱动方法类似,这些源空间ICA方法的实现需要修改以适应EEG/MEG数据的性质。例如,EEG/MEG中的ICA通常应用于对源数据进行奇异值分解(SVD)获得的时域子空间数据,该数据捕获了数据中的主要时域变化。因此,相对于fMRI数据上的空域ICA,EEG/MEG上的ICA通常被称为时域ICA。这种差异背后的主要数学原理是,假定EEG/MEG数据在时域上是统计独立的,而fMRI数据在空域上是统计独立的。此外,像ICA这样的数据驱动模式识别算法通常需要更多的样本。然而,由于从传感域到源域的逆映射,EEG/MEG源的空间维数急剧增加,因此,有研究报告可以在EEG/MEG数据上使用空域和时域ICAs,特别是当可以对时域数据进行降维时(例如使用主成分分析(PCA))。
有研究人员提出了一组混合方法,即结合基于种子的相关方法和时域ICA方法来表征EEG数据中的大脑网络。首先通过数据驱动方法(即ICA)从EEG静息态数据中识别出一组同时具有空间和时间模式的成分,即ICs。然后对所选IC时程与单个体素时程进行相关分析,利用相关层析成像来表征每个IC背后的大脑网络(图3C),以便于进行进一步的统计分析,因为相关值具有统计学意义,而IC权重没有。
在基础神经科学中,利用EEG/MEG数据研究大脑网络主要是为了回答以下三个方面的关键问题。
第一个关键问题是,在fMRI数据中识别出的类似大脑网络是否也能在EEG/MEG数据中检测到。在这里,相似性主要通过它们的空间模式来定义的,即覆盖相似分布的大脑区域。此外,如果可以同时记录多模态数据,例如同步记录的EEG-fMRI,则可以研究时域的相似性。与EEG相比,MEG具有相对较高的空间采样密度,首先利用MEG研究了基于电生理数据的源级脑网络的空间模式。有研究发现,MEG衍生的脑网络与fMRI衍生的脑网络大体一致(图4)。总而言之,目前已证实在fMRI数据中识别出的类似大脑网络也能在EEG/MEG数据中检测到,并且具有统计学上显著的高空间和时间相似性。
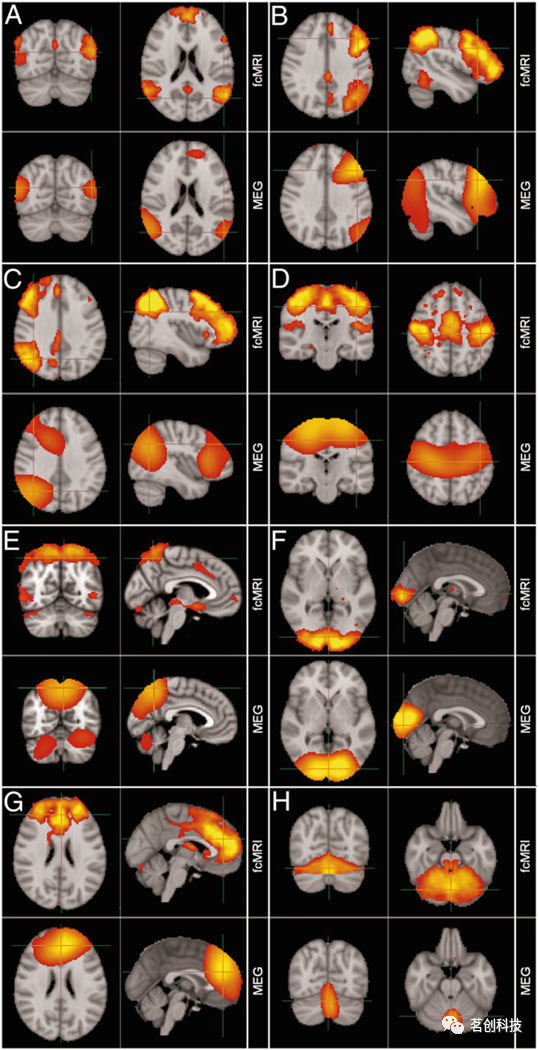
图4.与fMRI的ICNs相比,特定频段MEG的ICNs具有相似的空间映射,且均采用ICA方法。
第二个关键问题是,EEG/MEG数据(由于其毫秒级的时间分辨率)是否能比fMRI数据阐明更多有关大脑网络的信息。因此,EEG/MEG数据有望在更快的时间尺度上探测大脑网络的动态配置或重新配置,以及内在大脑网络的振荡节奏特征。在EEG和MEG脑网络研究中,已经报告了各种ICN的频率依赖空间模式。这些研究表明,内在的大脑网络具有频率依赖性,未来需要更多的研究来阐明它们的动态及其动态与频率分量之间的关系。
最后一个关键问题是,这些网络及其动态是否因大脑功能的变化而改变,例如在发育和衰老过程中。例如,Cohen等人(2013)分析了知觉辨别任务中记录的EEG数据,并证明了多个大规模脑网络(即内侧额叶和枕中枢)在不同频段(即theta和theta-alpha耦合)上以特定方向进行动态交互,从而支持感知决策过程中的灵活行为适应。Schafer等人(2014)利用MEG数据研究了内在脑网络的年龄依赖性变化,并揭示了各种脑网络内部和之间振荡通信的典型成熟模式(图5)。所有这些研究都清楚地表明,EEG和MEG是探测ICN特征的强大替代方法,并有助于更好地理解各种认知功能和行为背后的神经机制。
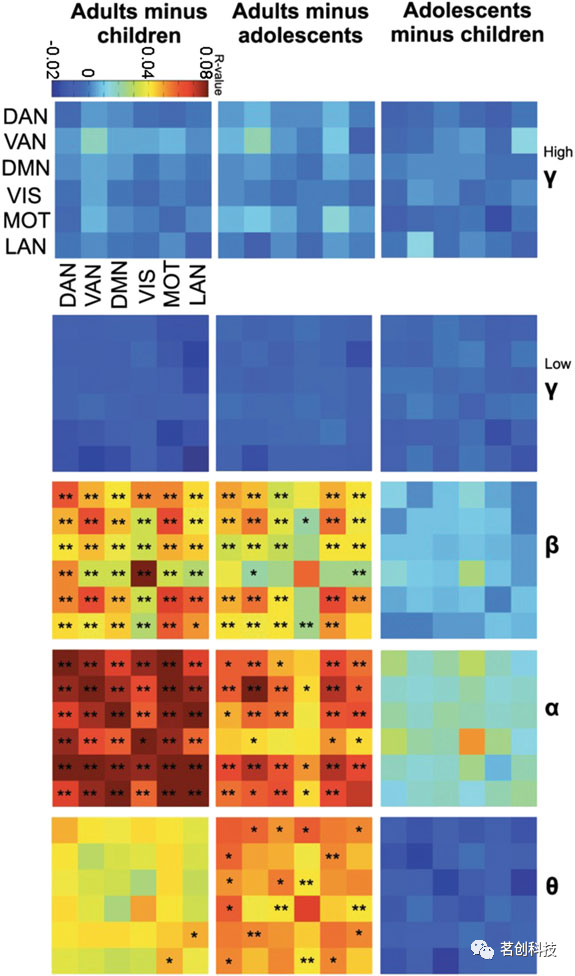
图5.五个频段上的网络连接在三个年龄组(儿童、青少年和成人)之间的对比:theta(4-7Hz)、alpha(8-14Hz)、beta(15-30Hz)、低频gamma(30-80Hz)和高频gamma(80-150Hz)。
使用其他神经成像模式的脑网络映射
颅内电生理记录-ECoG
颅内电生理记录是通过将电极直接植入人脑皮层或大脑深部获得。其中,ECoG是最常用的形式,用于测量人脑皮层的电生理信号。因此,ECoG是一种侵入性记录技术,空间覆盖范围有限,仅适用于特定患者(如癫痫患者)。由于ECoG测量颅骨下方的信号,因而其记录的局部大脑区域产生的电信号具有更高的信噪比和更宽的频率范围(高达几百赫兹,而EEG的频率则低于100Hz,因为头骨充当了一个低通滤波器)。因此,它可以被视为研究特定大脑网络的“黄金标准”,例如DMN或大脑网络的单个节点。不幸的是,由于ECoG的侵入性和有限的空间覆盖范围,通常不能用于同时研究多个大脑网络。
正电子发射断层扫描(PET)
PET是一种3D神经成像工具,与fMRI一样都能提供人脑体积图像。PET测量外源放射性标记化学剂的分布,这些化学剂指示整个人脑新陈代谢的变化。因此,基于PET的连接分析在文献中被称为代谢连接分析,并且在探索代谢脑网络方面有很长的历史。它可以将区域代谢信号变化与其他变化(例如,脑血流量(CBF)或耗氧量)分开,而这些变化在fMRI信号中是结合在一起的,因此难以完全分离。事实上,“功能连接”这个术语早在fMRI之前就已经在PET数据上进行了定义,而DMN最早是由Raichle等人(2001)使用PET发现的(图6)。由于PET的测量目标,已知代谢变化的脑疾病(如阿尔茨海默病)可以通过PET更好地评估脑网络的变化。然而,PET受其侵入性(即使用放射性剂)和有限的时间分辨率(即分钟级)的影响。作为主要的3D体积神经成像技术之一,与fMRI相比,其空间分辨率也相对较低。
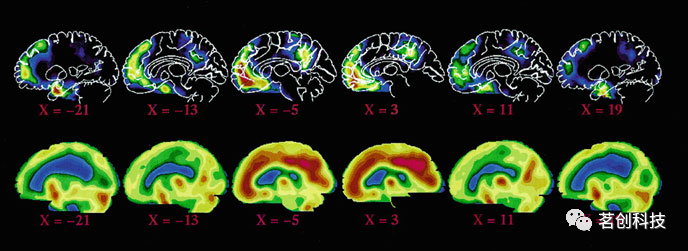
图6.首次使用PET数据识别出的DMN。
功能近红外光谱(fNIRS)
fNIRS是一种利用近红外光测量皮层组织中含氧血红蛋白(HbO)和脱氧血红蛋白(HbR)浓度变化的神经成像方法。fNIRS已被有效地用于表征成人和婴儿的静息态大脑网络,以及用于评估脑部疾病患者的异常脑网络。fNIRS同时测量HbO和HbR信号,而fMRI仅对HbR信号敏感。fNIRS通常具有比fMRI(~1Hz)更高的时间采样率(~10Hz),这提供了排除非神经元相关血流动力学变化的优势,例如由于呼吸和心跳引起的变化。此外,fNIRS更经济高效和具有便携性,可用于不适合使用MRI扫描仪的患者和婴儿等人群,以及具有挑战性的环境,如床旁脑电监测。此外,由于其相对较高的采样率,fNIRS揭示了人脑中频率特异性的功能连接。因此,fNIRS是fMRI在研究人类内在脑网络的一种补充选择。
使用同步多模态数据的脑网络映射
大脑网络已通过各种神经成像模式的数据进行识别和表征,这些神经成像模式中的每一种都提供了有关所研究网络的宝贵信息。从不同神经成像模式所获得的脑网络信息通常是重叠的,但也互为补充。因此,整合来自多种神经成像模式的数据有望提供更全面的内在大脑网络视图。整合来自不同神经成像模式的信息,主要有三类方法:①融合来自多种神经成像研究的关于脑网络的发现;②融合来自多种神经成像模式的数据,以重建和探测大脑网络;③通过调整脑网络计算神经模型的参数来模拟多种神经成像模式的数据以进行研究。第一类方法是对各类神经成像数据研究的总体发现进行高水平的整合,以绘制有关大脑网络的全面图像。第二类是直接融合来自多个神经成像模式的数据,可以分别记录或同时记录来自同一个人的数据。由于数据性质的差异,例如空间和时间尺度以及不同神经生理量的表征,通常需要新的信号处理或数据分析方法将它们融合在一起,以识别大脑网络的模式或研究其特征。最后一类侧重于大脑网络的计算神经模型,而不是映射大脑网络。接下来,本文介绍了三种同步记录的多模态脑网络研究,即EEG-fMRI,EEG-MEG和PET-MRI/PET-MRI-EEG。
同步EEG-fMRI
如前所述,已分别使用fMRI或EEG/MEG数据对大脑网络进行了广泛的研究,并在这两种成像方式之间发现了一致的脑网络。然而,单独的记录具有局限性,因为这些研究中的数据不是同时记录的,许多甚至不是来自同一个人。从技术上讲,MEG尚不能与fMRI一起使用,而EEG信号已被广泛证明可以在MRI扫描仪中记录。这种能力为同时分析和整合电信号和血流动力学信号提供了机会。据信,从同步EEG-fMRI数据中识别出的大脑网络将更加完整,因为EEG和fMRI从不同的潜在生理过程中提供了关于大脑网络的互补信息。同时使用EEG和fMRI成像的一个明显好处是,在研究大脑网络时,既具有高时间分辨率(由EEG提供)又具有高空间分辨率(由fMRI提供)。这里有一个很好的例子来说明同时进行EEG-fMRI研究的好处。实时fMRI神经反馈首次被证明可有效降低PTSD患者的临床症状评分。多模态EEG-fMRI数据与机器学习策略相结合,随后发现了EEG指纹,从而导致有效的单独EEG神经反馈,以减轻PTSD患者的临床症状。
同步EEG-fMRI数据已被用于研究fMRI衍生的脑网络在空域和时域中的电生理相关性。这类研究之所以变得重要,是因为fMRI可以测量次级神经反应,但同时它也是映射大脑网络的主要神经成像方式。虽然空间特征也可以在单独的EEG和fMRI记录中进行研究,但时间特征只能在同步记录的数据中研究。在这方面,EEG动态的时间特征(其形式为窄带功率、全带功率或高功率状态下的瞬态电位地形图),在至少部分与fMRI衍生的大脑网络重叠的脑区中被发现。有研究人员考察了健康个体和脑疾病患者的多个内在脑网络的EEG电生理相关性。这些数据表明,瞬时静息态EEG地形通过神经血管耦合与静息期fMRI波动在时间上相关。更重要的是,通过对高密度EEG进行电生理源成像,结果发现了EEG ICNs的皮层重建与许多fMRI ICNs具有一对一的对应关系(图7),包括视觉、感觉运动和听觉网络,以及默认模式网络、注意和语言网络。
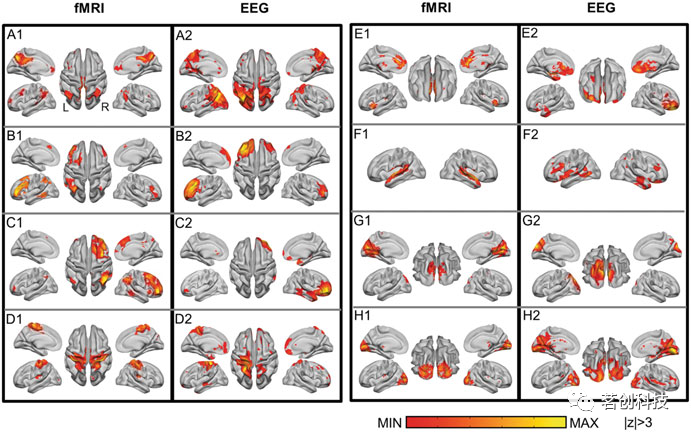
图7.八对一对一匹配的fMRI ICNs和EEG ICNs:(A)默认模式网络(DMN);(B)额顶叶(左);(C)额顶叶(右);(D)感觉运动;(E)执行控制;(F)听觉;(G)视觉(内侧);(H)视觉(外侧)。所有空间地图的阈值均为|z|=3。
同步EEG-MEG
EEG和MEG是两种广泛用于研究大脑网络的神经成像模式。由于它们是由相同的电信号源产生的,因此它们之间的差异并不像EEG和fMRI之间的差异那么大,而是由于信号产生机制的不同而有所不同。EEG是由于大脑中电信号源的被动传导,而MEG显示了与其耦合的时变磁场变化。每种模式都有其自身的优点和缺点。MEG对切向头部表面的脑源更敏感,更善于捕捉短距离功能连接。EEG对径向头部表面的脑源更敏感,并且能更好地捕获远距离功能连接。此外,与EEG相比,MEG受容积传导效应的影响较小。使用同步EEG-MEG研究脑网络主要有两个目标:①MEG在揭示大脑网络方面具有优越的性能,但由于技术的复杂性和高昂的成本,其仪器的可用性较差,因此,如果能发现EEG具有与MEG相似的性能,则可以用EEG取代MEG,并拓宽临床应用范围;②EEG和MEG的整合,可以比单独的EEG或MEG揭示更多、更好的脑网络知识,因为两者具有互补信号优势。EEG和MEG的组合已被广泛用于皮质源成像,这是研究EEG/MEG源域脑网络的关键步骤。在模拟和实验数据中的研究均发现,与单独使用EEG或MEG相比,EEG和MEG联合使用时的皮质源重建性能明显提高。
同步PET-MRI和PET-MRI-EEG
自2008年首次证明同步PET-MRI在人脑上的可行性以来,已经有大量研究使用同步PET-MRI来揭示人脑激活和功能连接。PET和MRI的结合可以同时测量多种生物学特性,例如,测量同一个体在完全相同的生理条件下的葡萄糖代谢(PET)和耗氧量(fMRI)。从技术上讲,两种模式所记录的体积数据帧可以在空域和时域中完美地共配准。Savio等人(2017)对健康参与者的同步PET-MRI数据使用ICA方法,发现两种模式都可以检测到相似的ICNs。由于PET对疾病条件下发生的局部代谢异常很敏感,通常选择PET中的异常热点作为种子区域,用fMRI进行交叉模态相关分析,以映射异常的脑网络。这进一步表明,同步PET-MRI是识别脑部疾病患者异常脑网络的一种很有前途的工具。
目前,PET-MRI已进一步与EEG集成,可同时生成三模态PET-MRI-EEG测量结果,从而以毫秒级的时间分辨率添加了映射ICNs的电生理数据,这是PET或fMRI所无法提供的。通过解决许多技术挑战,来自健康被试DMN的互补指纹和精神分裂症患者的潜在脑网络生物标志物已被报告使用了这种新型的综合神经成像方式。更有趣的是,三模态数据进一步表明,EEG微状态的神经元生成器和fMRI测量的脑网络(例如,区域同质性,中心性程度等)与GABAA的神经受体结合以及葡萄糖代谢的关系要比mGluR5紧密得多,这表明抑制过程是静息态ICN的核心机制。基于此,需要注意的是,多种神经成像模式与同步记录的组合不仅可以同时揭示ICNs的多个方面,还可以产生无法在单独记录中阐明的有关ICNs的新知识。
整合结构和功能数据的脑网络映射
值得注意的是,从功能性神经成像数据识别大脑网络时,通常需要大脑结构数据,如结构MRI。脑网络也可以纯粹基于大脑结构数据进行研究,例如,弥散张量成像(DTI)数据,称为结构脑网络。接下来将讨论这两种类型的结构性大脑数据及其在脑网络研究中的使用情况。
结构MRI数据提供了从功能数据中识别出的脑网络的解剖信息。根据功能数据类型的不同,结构MRI数据的使用也不同。对于直接在人脑体素上测量的功能数据,即fMRI和PET数据,结构MRI数据与功能数据在空间上进行配准,并作为可视化功能数据热点的解剖学基础,提供神经激活的结构定位和已识别的大脑网络(即ICNs)。对于在头部表面测量的功能数据,如EEG/MEG和fNIRS,结构MRI数据用于构建脑源和体积导体模型,以解决逆源重建问题。在这里,功能数据的传感器位置需要与结构MRI数据的模型进行共配准,共配准的准确性会对逆源重建的精度产生很大影响。建议使用个人的结构MRI数据来确定模型的准确性和共配准,尽管这样做会导致实验成本和数据分析工作量的增加。此外,在涉及组水平比较的研究中,需要将单个结构数据及其派生模型映射到标准模板,以便进行组水平分析和统计推断,例如,MNI ICBM152大脑3D体积数据和来自FREESURFER的大脑皮层表面模型(https://surfer.nmr.mgh.harvard.edu/)。
DTI也可以直接用于映射大脑结构网络。DTI测量水分子在大脑中的扩散,使用单个volume体素中的不同扩散速度来推断白质束穿过体素的方向,并通过将所有体素的这些信息聚合在一起来重建纤维束。据信,功能连接的背后是白质纤维的物理路径,而相比于时变功能连接,结构连接在更长的时间尺度上是相对固定的。同时,DTI为研究不同ICNs的结构和功能连接之间的关系提供了重要手段。在许多大规模ICNs研究中已经使用了结构-功能融合,例如图8所示的DMN和感觉运动网络。在神经退行性疾病研究中也报告了这种融合的结构和功能连接之间的相应变化。

图8.在DMN中,fMRI衍生的功能连接与DTI衍生的结构连接一致。
结论
综上所述,脑网络映射是一个非常活跃的研究领域。在进行脑网络映射研究中已经建立了许多神经成像技术和模式。特别是,内在大脑网络的发现及其空间、时间、频谱和动态模式的特征是这些研究活动最重要的发展成果。这些发现解决了基础和临床神经科学中许多具有挑战性的问题。有关该领域的研究将在未来继续蓬勃发展。这不仅涉及开发新的工具(包括仪器和分析方法)以更好地映射人脑网络,而且还将揭示有关人脑如何工作的新知识。此外,在临床研究中,脑网络研究的发展有望显著提高我们诊断和治疗各种脑部疾病的能力,包括AD、PTSD、ASD和许多其他脑部疾病。
参考文献(上下滑动查看):
Biswal, B., Yetkin, F.Z., Haughton, V.M., Hyde, J.S.: Functional connectivity in the motor cortex of resting human brain using echo-planar MRI. Magn. Reson. Med. 34(4), 537-541(1995). https://doi.org/10.1002/mrm.1910340409
Raichle, M.E., MacLeod, A.M., Snyder, A.Z., Powers, W.J., Gusnard, D.A., Shulman, G.L.: A default mode of brain function. Proc. Natl. Acad. Sci. U. S. A. 98(2), 676-682 (2001). https://doi.org/10.1073/pnas.98.2.676
Damoiseaux, J.S., Rombouts, S.A., Barkhof, F., Scheltens, P., Stam, C.J., Smith, S.M., Beckmann, C.F.: Consistent resting-state networks across healthy subjects. Proc. Natl. Acad. Sci. U. S. A. 103(37), 13848-13853 (2006). https://doi.org/10.1073/pnas.0601417103
Fox, M.D., Raichle, M.E.: Spontaneous fluctuations in brain activity observed with functional magnetic resonance imaging. Nat. Rev. Neurosci. 8(9), 700-711 (2007). https://doi.org/10.1038/nrn2201
Varela, F., Lachaux, J.P., Rodriguez, E., Martinerie, J.: The brainweb: phase synchronization and large-scale integration. Nat. Rev. Neurosci. 2(4), 229-239 (2001). https://doi.org/10.1038/35067550
Kenet, T., Bibitchkov, D., Tsodyks, M., Grinvald, A., Arieli, A.: Spontaneously emerging cortical representations of visual attributes. Nature. 425(6961), 954-956 (2003). https://doi.org/10.1038/nature02078
Raichle, M.E., Mintun, M.A.: Brain work and brain imaging. Annu. Rev. Neurosci. 29, 449-476 (2006). https://doi.org/10.1146/annurev.neuro.29.051605.112819
Raichle, M.E., Snyder, A.Z.: A default mode of brain function: a brief history of an evolving idea. NeuroImage. 37(4), 1083–1090.; discussion 1097-1089 (2007). https://doi.org/10.1016/j.neuroimage.2007.02.041
Fox, M.D., Snyder, A.Z., Vincent, J.L., Corbetta, M., Van Essen, D.C., Raichle, M.E.:
The human brain is intrinsically organized into dynamic, anticorrelated functional networks. Proc. Natl. Acad. Sci. U. S. A. 102(27), 9673-9678 (2005). https://doi.org/10.1073/pnas.0504136102
Jafri, M.J., Pearlson, G.D., Stevens, M., Calhoun, V.D.: A method for functional network connectivity among spatially independent resting-state components in schizophrenia. NeuroImage. 39(4), 1666-1681 (2008). https://doi.org/10.1016/j.neuroimage.2007.11.001
Luckhoo, H., Hale, J.R., Stokes, M.G., Nobre, A.C., Morris, P.G., Brookes, M.J., Woolrich, M.W.: Inferring task-related networks using independent component analysis in magnetoencephalography. NeuroImage. 62(1), 530–541 (2012). https://doi.org/10.1016/j.neuroimage.2012.04.046
Smith, S.M., Fox, P.T., Miller, K.L., Glahn, D.C., Fox, P.M., Mackay, C.E., Filippini, N., Watkins, K.E., Toro, R., Laird, A.R., Beckmann, C.F.: Correspondence of the brain’s functional architecture during activation and rest. Proc. Natl. Acad. Sci. U. S. A. 106(31), 13040–13045 (2009). https://doi.org/10.1073/pnas.0905267106
Zhang, N., Rane, P., Huang, W., Liang, Z., Kennedy, D., Frazier, J.A., King, J.: Mapping resting-state brain networks in conscious animals. J. Neurosci. Methods. 189(2), 186–196(2010). https://doi.org/10.1016/j.jneumeth.2010.04.001
Vincent, J.L., Patel, G.H., Fox, M.D., Snyder, A.Z., Baker, J.T., Van Essen, D.C., Zempel, J.M., Snyder, L.H., Corbetta, M., Raichle, M.E.: Intrinsic functional architecture in the anaesthetized monkey brain. Nature. 447(7140), 83–86 (2007). https://doi.org/10.1038/nature05758
Fox, M.D., Snyder, A.Z., Zacks, J.M., Raichle, M.E.: Coherent spontaneous activity accounts for trial-to-trial variability in human evoked brain responses. Nat. Neurosci. 9(1), 23–25(2006). https://doi.org/10.1038/nn1616
Fox, M.D., Snyder, A.Z., Vincent, J.L., Raichle, M.E.: Intrinsic fluctuations within cortical systems account for intertrial variability in human behavior. Neuron. 56(1), 171–184 (2007). https://doi.org/10.1016/j.neuron.2007.08.023
Mason, M.F., Norton, M.I., Van Horn, J.D., Wegner, D.M., Grafton, S.T., Macrae, C.N.: Wandering minds: the default network and stimulus-independent thought. Science. 315(5810), 393–395 (2007). https://doi.org/10.1126/science.1131295
Buckner, R.L., Andrews-Hanna, J.R., Schacter, D.L.: The brain’s default network: anatomy, function, and relevance to disease. Ann. N. Y. Acad. Sci. 1124, 1–38 (2008). https://doi.org/10.1196/annals.1440.011
Douw, L., van Dellen, E., Gouw, A.A., Griffa, A., de Haan, W., van den Heuvel, M.,
Hillebrand, A., Van Mieghem, P., Nissen, I.A., Otte, W.M., Reijmer, Y.D., Schoonheim, M.M., Senden, M., van Straaten, E.C.W., Tijms, B.M., Tewarie, P., Stam, C.J.: The road ahead in clinical network neuroscience. Netw Neurosci. 3(4), 969–993 (2019). https://doi.org/10.1162/netn_a_00103
Fox, M.D., Buckner, R.L., Liu, H., Chakravarty, M.M., Lozano, A.M., Pascual-Leone,
A.: Resting-state networks link invasive and noninvasive brain stimulation across diverse psychiatric and neurological diseases. Proc. Natl. Acad. Sci. U. S. A. 111(41), E4367–E4375(2014). https://doi.org/10.1073/pnas.1405003111
Fox, M.D., Greicius, M.: Clinical applications of resting state functional connectivity. Front. Syst. Neurosci. 4, 19 (2010). https://doi.org/10.3389/fnsys.2010.00019
Stam, C.J.: Modern network science of neurological disorders. Nat. Rev. Neurosci. 15(10), 683–695 (2014). https://doi.org/10.1038/nrn3801
Tost, H., Bilek, E., Meyer-Lindenberg, A.: Brain connectivity in psychiatric imaging genetics. NeuroImage. 62(4), 2250–2260 (2012). https://doi.org/10.1016/j.neuroimage.2011.11.007
Zhang, D., Raichle, M.E.: Disease and the brain’s dark energy. Nat. Rev. Neurol. 6(1), 15–28(2010). https://doi.org/10.1038/nrneurol.2009.198
Aiello, M., Cavaliere, C., Salvatore, M.: Hybrid PET/MR imaging and brain connectivity. Front. Neurosci. 10, 64 (2016). https://doi.org/10.3389/fnins.2016.00064
Bassett, D.S., Bullmore, E.T.: Human brain networks in health and disease. Curr. Opin. Neurol. 22(4), 340–347 (2009). https://doi.org/10.1097/WCO.0b013e32832d93dd
Stam, C.J.: Use of magnetoencephalography (MEG) to study functional brain networks
in neurodegenerative disorders. J. Neurol. Sci. 289(1-2), 128–134 (2010). https://doi.org/10.1016/j.jns.2009.08.028
Nugent, A.C., Robinson, S.E., Coppola, R., Furey, M.L., Zarate Jr., C.A.: Group differences in MEG-ICA derived resting state networks: application to major depressive disorder. NeuroImage. 118, 1–12 (2015). https://doi.org/10.1016/j.neuroimage.2015.05.051
Drysdale, A.T., Grosenick, L., Downar, J., Dunlop, K., Mansouri, F., Meng, Y., Fetcho, R.N., Zebley, B., Oathes, D.J., Etkin, A., Schatzberg, A.F., Sudheimer, K., Keller, J., Mayberg, H.S., Gunning, F.M., Alexopoulos, G.S., Fox, M.D., Pascual-Leone, A., Voss, H.U., Casey, B.J., Dubin, M.J., Liston, C.: Resting-state connectivity biomarkers define neurophysiological subtypes of depression. Nat. Med. 23(1), 28–38 (2017). https://doi.org/10.1038/nm.4246
Wang, L., Hermens, D.F., Hickie, I.B., Lagopoulos, J.: A systematic review of resting-state functional-MRI studies in major depression. J. Affect. Disord. 142(1-3), 6–12 (2012). https://doi.org/10.1016/j.jad.2012.04.013
Anand, A., Li, Y., Wang, Y., Wu, J., Gao, S., Bukhari, L., Mathews, V.P., Kalnin, A., Lowe, M.J.: Activity and connectivity of brain mood regulating circuit in depression: a functional magnetic resonance study. Biol. Psychiatry. 57(10), 1079–1088 (2005). https://doi.org/10.1016/j.biopsych.2005.02.021
Greicius, M.D., Flores, B.H., Menon, V., Glover, G.H., Solvason, H.B., Kenna, H., Reiss, A.L., Schatzberg, A.F.: Resting-state functional connectivity in major depression: abnormally increased contributions from subgenual cingulate cortex and thalamus. Biol. Psychiatry. 62(5), 429–437 (2007). https://doi.org/10.1016/j.biopsych.2006.09.020
Sheline, Y.I., Raichle, M.E.: Resting state functional connectivity in preclinical
Alzheimer’s disease. Biol. Psychiatry. 74(5), 340–347 (2013). https://doi.org/10.1016/j.biopsych.2012.11.028
Foo, H., Mather, K.A., Jiang, J., Thalamuthu, A., Wen, W., Sachdev, P.S.: Genetic influence on ageing-related changes in resting-state brain functional networks in healthy adults: a systematic review. Neurosci. Biobehav. Rev. 113, 98–110 (2020). https://doi.org/10.1016/j.neubiorev.2020.03.011
Dennis, E.L., Thompson, P.M.: Functional brain connectivity using fMRI in aging and
Alzheimer’s disease. Neuropsychol. Rev. 24(1), 49–62 (2014). https://doi.org/10.1007/s11065-014-9249-6
Mandal, P.K., Banerjee, A., Tripathi, M., Sharma, A.: A comprehensive review of
magnetoencephalography (MEG) studies for brain functionality in healthy aging and
Alzheimer’s Disease (AD). Front. Comput. Neurosci. 12, 60 (2018). https://doi.org/10.3389/fncom.2018.00060
Greicius, M.D., Srivastava, G., Reiss, A.L., Menon, V.: Default-mode network activity distinguishes Alzheimer’s Disease from healthy aging: evidence from functional MRI. Proc. Natl. Acad. Sci. U. S. A. 101(13), 4637–4642 (2004). https://doi.org/10.1073/pnas.0308627101
Buckner, R.L., Snyder, A.Z., Shannon, B.J., LaRossa, G., Sachs, R., Fotenos, A.F., Sheline, Y.I., Klunk, W.E., Mathis, C.A., Morris, J.C., Mintun, M.A.: Molecular, structural, and functional characterization of Alzheimer’s Disease: evidence for a relationship between default activity, amyloid, and memory. J. Neurosci. 25(34), 7709–7717 (2005). https://doi.org/10.1523/JNEUROSCI.2177-05.2005
Del Guerra, A., Ahmad, S., Avram, M., Belcari, N., Berneking, A., Biagi, L., Bisogni, M.G., Brandl, F., Cabello, J., Camarlinghi, N., Cerello, P., Choi, C.H., Coli, S., Colpo, S., Fleury, J., Gagliardi, V., Giraudo, G., Heekeren, K., Kawohl, W., Kostou, T., Lefaucheur, J.L., Lerche, C., Loudos, G., Morrocchi, M., Muller, J., Mustafa, M., Neuner, I., Papadimitroulas, P., Pennazio, F., Rajkumar, R., Brambilla, C.R., Rivoire, J., Kops, E.R., Scheins, J., Schimpf, R., Shah, N.J., Sorg, C., Sportelli, G., Tosetti, M., Trinchero, R., Wyss, C., Ziegler, S., Consortium, T.: TRIMAGE: a dedicated trimodality (PET/MR/EEG) imaging tool for schizophrenia. Eur Psychiatry. 50, 7–20 (2018). https://doi.org/10.1016/j.eurpsy.2017.11.007
Whitfield-Gabrieli, S., Thermenos, H.W., Milanovic, S., Tsuang, M.T., Faraone, S.V., McCarley, R.W., Shenton, M.E., Green, A.I., Nieto-Castanon, A., LaViolette, P., Wojcik, J., Gabrieli, J.D., Seidman, L.J.: Hyperactivity and hyperconnectivity of the default network in schizophrenia and in first-degree relatives of persons with schizophrenia. Proc. Natl. Acad. Sci. U. S. A. 106(4), 1279–1284 (2009). https://doi.org/10.1073/pnas.0809141106
Lynall, M.E., Bassett, D.S., Kerwin, R., McKenna, P.J., Kitzbichler, M., Muller, U., Bullmore, E.: Functional connectivity and brain networks in schizophrenia. J. Neurosci. 30(28), 9477-9487 (2010). https://doi.org/10.1523/JNEUROSCI.0333-10.2010
Calhoun, V.D., Eichele, T., Pearlson, G.: Functional brain networks in schizophrenia: a review. Front. Hum. Neurosci. 3, 17 (2009). https://doi.org/10.3389/neuro.09.017.2009
Jalili, M., Knyazeva, M.G.: EEG-based functional networks in schizophrenia. Comput. Biol. Med. 41(12), 1178-1186 (2011). https://doi.org/10.1016/j.compbiomed.2011.05.004
Li, S., Hu, N., Zhang, W., Tao, B., Dai, J., Gong, Y., Tan, Y., Cai, D., Lui, S.: Dysconnectivity of multiple brain networks in schizophrenia: a meta-analysis of resting-state functional connectivity. Front Psychiatry. 10, 482 (2019). https://doi.org/10.3389/fpsyt.2019.00482
Savio, A., Funger, S., Tahmasian, M., Rachakonda, S., Manoliu, A., Sorg, C., Grimmer, T., Calhoun, V., Drzezga, A., Riedl, V., Yakushev, I.: Resting-state networks as simultaneously measured with functional MRI and PET. J. Nucl. Med. 58(8), 1314-1317 (2017). https://doi.org/10.2967/jnumed.116.185835
Cohen, M.X., van Gaal, S.: Dynamic interactions between large-scale brain networks predict behavioral adaptation after perceptual errors. Cereb. Cortex. 23(5), 1061-1072 (2013). https://doi.org/10.1093/cercor/bhs069
Schafer, C.B., Morgan, B.R., Ye, A.X., Taylor, M.J., Doesburg, S.M.: Oscillations, networks, and their development: MEG connectivity changes with age. Hum. Brain Mapp. 35(10), 5249-5261 (2014). https://doi.org/10.1002/hbm.22547
Yuan, H., Zotev, V., Phillips, R., Drevets, W.C., Bodurka, J.: Spatiotemporal dynamics of the brain at rest–exploring EEG microstates as electrophysiological signatures of BOLD resting state networks. NeuroImage. 60(4), 2062–2072 (2012). https://doi.org/10.1016/j.neuroimage.2012.02.031
Britz, J., Van De Ville, D., Michel, C.M.: BOLD correlates of EEG topography reveal
rapid resting-state network dynamics. NeuroImage. 52(4), 1162–1170 (2010). https://doi.org/10.1016/j.neuroimage.2010.02.052
