与转录组结合,开发下一代诊断技术,或许是医学AI领域的下一个热点|个人观点·24-09-21
小罗碎碎念
观点分享:科研本身是一件枯燥的事情,所以我们尽可能的去寻找一些同伴,也许前路的风景又会焕然一新。
今天所有的推文都围绕一个人展开——Faisal Mahmood。说实话,今天的状态并不好,写推文的感觉很不对,但是我知道,我今天不写,也许下次再想起来,可能就是几个月以后的事了。每天都觉得时间不够用,可回头好像又不知道时间去哪里了。
今天的四篇文章,分别从以下几个角度探索了病理AI的未来发展方向:
- 3D病理研究
- 与转录组数据结合(这也是我前两天分析张泽民院士的原因)
- 研究下一代诊断病理技术,然后再与AI结合
个人认为,病理AI的方向从2021年的CLAM开始,就一直被Faisal Mahmood组引领,直至2024年,Cell和Nature都刊登过他们课题组的文章——从3D病理到通用模型。
目前发表在顶刊的大模型,作者大都来自哈佛大学、斯坦福大学和华盛顿大学。所以,我也在思考一个问题,为什么引领行业的研究会率先在国外开展?
昨天晚上睡觉前在翻阅Faisal Mahmood相关的资料,看到他的社交平台基本都是学术动态,我就在想,在国外和国内开展研究,区别到底有多大?在国外成长的几年,能否在国内找到替代的方式——是否只要语言能力和视野得到同样的提升就可以?(国内的头部课题组拥有的条件和氛围是可以和国外媲美的)
PS:其实,归到最后,还是要看自己选择的老板如何,以及自己的能力如何,否则去哪都一样。
一、2.5D多实例学习:优化病理学家的3D病理数据评估
这篇文章介绍了CARP3D,一种基于2.5D多实例学习的深度学习方法,用于从3D病理数据中筛选出高风险的2D切片,以提高病理学家的诊断效率和准确性。

一作&通讯
| 作者角色 | 作者姓名 | 单位名称(英文) | 单位名称(中文) |
|---|---|---|---|
| 第一作者 | Gan Gao | University of Washington | 华盛顿大学 |
| 通讯作者 | Andrew H. Song | Mass General Brigham, Harvard University | 麻省总医院,哈佛大学 |
| 通讯作者 | Jonathan T.C. Liu | University of Washington | 华盛顿大学 |
这篇文章是关于一种名为CARP3D的深度学习方法,它用于从3D病理数据中筛选出最具风险的2D切片,以指导病理学家进行评估。
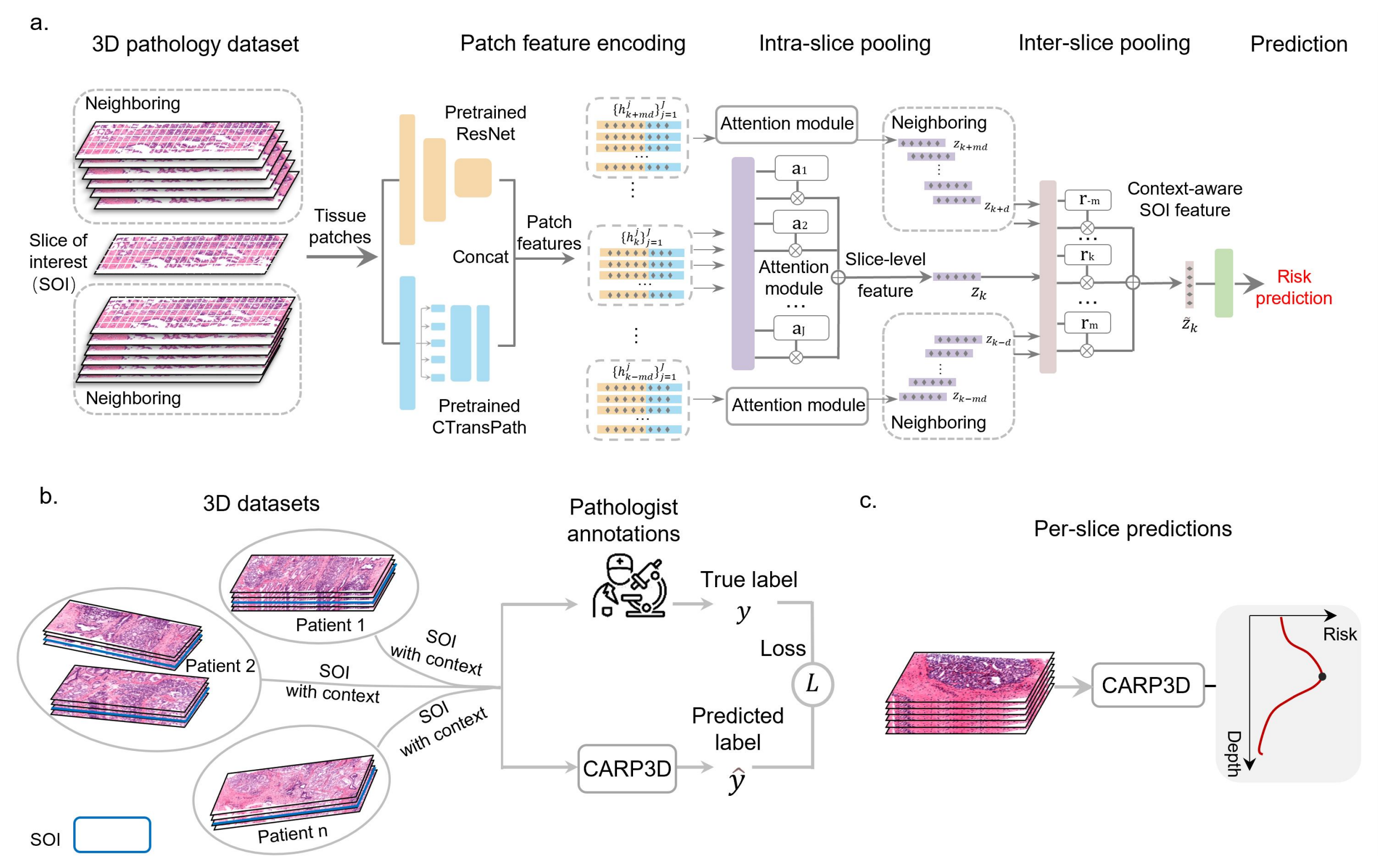
a) 2D切片编码与特征聚合:
- 切片编码: 目标切片(SOI)及其邻近切片使用预训练的ResNet50和CTransPath模型进行编码。这些模型能够从每个小块(patch)中提取特征。
- 特征聚合: 通过一个内部切片注意力模块(intra-slice attention module),将每个切片内小块级别的特征聚合成切片级别的特征。同时,通过一个跨切片池化模块(inter-slice pooling module),将邻近切片的特征聚合起来,形成具有上下文感知的目标切片特征,用于后续的风险预测。
b) 训练阶段:
- 在训练过程中,从训练集的每个3D样本中选择切片,CARP3D模型学习预测病理学家提供的地面真实标签(ground truth labels)。这意味着模型通过这些标签来学习区分高风险和低风险的切片。
c) 模型部署与风险评估:
- 在3D病理数据上部署CARP3D模型,进行逐切片的风险评估。模型会识别出风险最高的切片,以供病理学家进行进一步的审查。这有助于病理学家集中注意力于最有可能包含重要病理信息的切片,从而提高诊断的效率和准确性。
以下是文章的主要内容概括:
- 背景:传统的病理诊断依赖于病理学家对2D组织切片的评估,但这种方法可能无法全面代表3D组织样本。随着3D病理技术的发展,如开放顶部光片显微镜(OTLS),可以对组织进行非破坏性成像,提供了改善诊断的可能性。
- CARP3D方法:提出了一种基于深度学习的筛选方法,自动识别3D活检样本中风险最高的2D切片,以提高病理学家的审查效率。该方法通过在每个切片内进行基于注意力的聚合,然后对相邻切片进行池化,计算出上下文感知的2.5D风险评分。
- 实验:在前列腺癌风险分层方面,CARP3D在筛选切片方面达到了90.4%的AUC(曲线下面积),超过了仅依赖2D切片独立分析的方法(AUC=81.3%)。这表明整合额外的深度上下文信息可以增强模型的区分能力。
- 实验结果:通过在前列腺癌活检样本上的应用,展示了CARP3D在区分低级别和中高级别前列腺癌方面的有效性。使用2.5D方法比2D方法有显著的性能提升。
- 结论:CARP3D通过利用3D病理数据中的上下文信息,提高了病理学家诊断的准确性,并可能加速3D病理学的临床应用。
文章还讨论了相关工作,包括2D全幻灯片图像(WSI)分类的MIL框架、支持诊断决定的计算3D病理学,以及2.5D分析在3D数据中的应用。此外,文章还提供了实验设置、数据描述、实现细节、基线比较和结果分析。
二、转录组引导的全幻灯片图像表示学习
这篇文章介绍了一种名为TANGLE的新型自监督学习方法,通过结合基因表达数据和组织切片图像,提高了在计算病理学中对全幻灯片图像的表示学习能力。

一作&通讯
| 作者角色 | 作者姓名 | 单位名称(英文) | 单位名称(中文) |
|---|---|---|---|
| 第一作者 | Guillaume Jaume | Mass General Brigham, Harvard University | 麻省总医院,哈佛大学 |
| 通讯作者 | Drew F.K. Williamson | Emory University School of Medicine | 埃默里大学医学院 |
这篇文章是关于在计算病理学中使用转录组引导的幻灯片表示学习的研究。研究者们提出了一种名为TANGLE的多模态预训练策略,它结合了来自基因表达档案的信息和组织切片图像,以改善超大像素全幻灯片图像(WSIs)的学习。
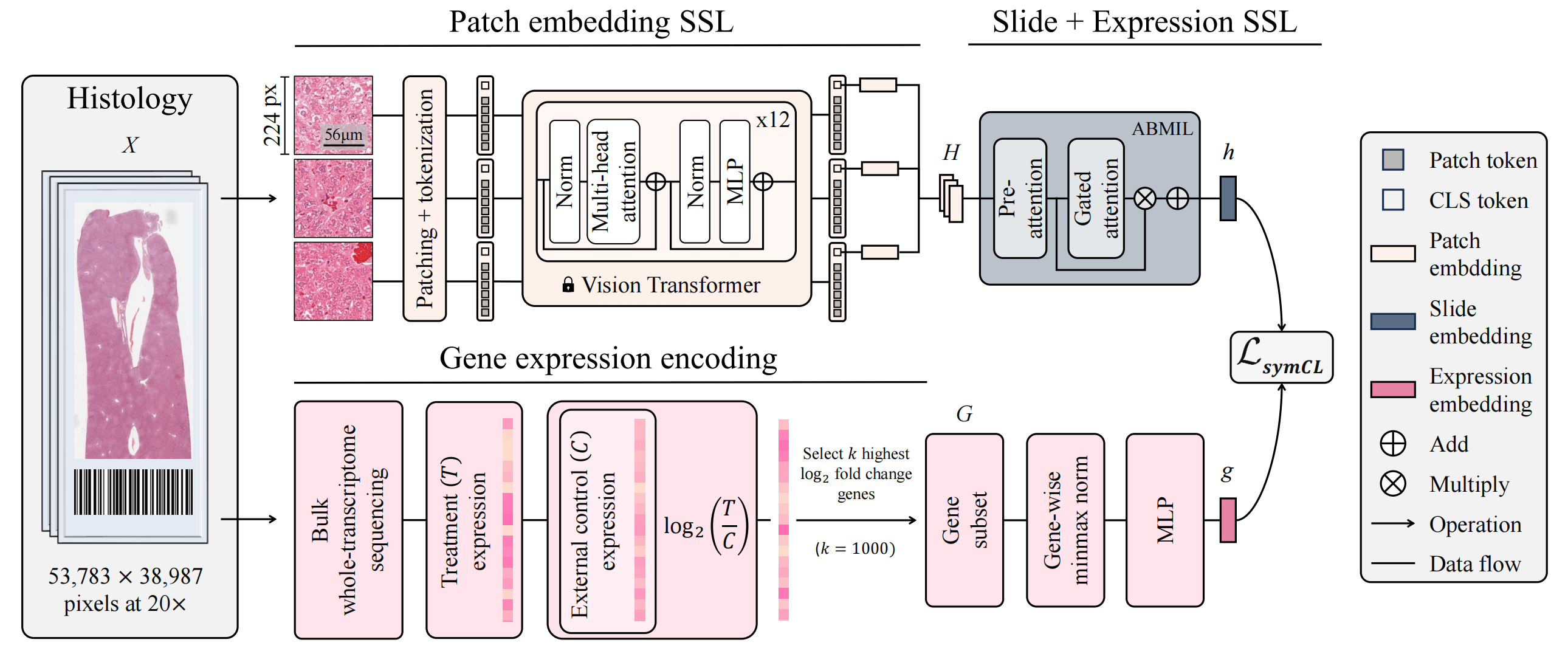
以下是对图中描述的流程的分析:
-
输入组织切片:输入的是一张待处理的组织学切片(Histology Slide)。
-
图像分块:将组织切片图像分割成小块(Patches),这是为了能够使用预训练的视觉编码器处理这些小块。
-
预训练视觉编码器:使用预训练的视觉编码器(Vision Encoder)对这些小块进行编码,得到对应的小块嵌入(Patch Embeddings)。
-
ABMIL模块:将小块嵌入输入到注意力机制的多示例学习(Attention-based Multiple Instance Learning, ABMIL)模块,该模块通过学习小块级别的注意力权重,将小块嵌入聚合成一个幻灯片嵌入(Slide Embedding)。
-
基因表达数据编码:同时,将对应的基因表达数据(Gene Expression Data)通过一个多层感知机(Multilayer Perceptron, MLP)进行编码,得到基因表达嵌入(Expression Embedding)。
-
对称对比目标:通过一个对称的对比学习目标(Symmetric Contrastive Objective, LsymCL),学习将两种模态(图像和基因表达)的嵌入进行对齐。
-
推理过程:在推理(Inference)阶段,将查询切片(Query Slide)通过视觉编码器和训练好的聚合模块(Pooling Module)编码成幻灯片嵌入,用于下游任务。
这个流程展示了TANGLE模型如何结合组织学图像和基因表达数据,通过自监督学习的方式进行预训练,以学习能够捕捉到组织学切片和基因表达数据之间复杂关系的嵌入表示。这种嵌入表示可以用于多种下游任务,如分类、检索等,而无需额外的标注信息。
文章的主要贡献包括:
-
TANGLE框架:利用特定于模态的编码器,通过对比学习对齐幻灯片和表达数据的输出,以学习幻灯片嵌入。
-
多模态对比学习:通过对称的对比学习目标,解决模态异质性问题,使幻灯片和表达嵌入对齐。
-
跨物种和器官的预训练:TANGLE在人类和大鼠的不同器官样本上进行了预训练,包括肝脏、乳腺和肺。
-
少样本学习性能:在独立的测试数据集上,TANGLE在少样本分类、原型分类和幻灯片检索任务上显示出比监督学习和自监督学习基线更好的性能。
-
代码开源:研究的代码已经在GitHub上公开。
-
实验和结果:使用TANGLE进行的实验表明,它在多种下游任务中都取得了显著的性能提升。
-
解释性分析:通过后处理解释性分析,研究者们能够从对齐的潜在空间中提取有关生物学过程的见解。
文章还讨论了与自监督视觉表示学习、计算病理学中的自监督学习、以及病理学中的监督学习相关的工作。此外,文章还介绍了TANGLE的具体方法,包括幻灯片编码器、基因表达编码器和多模态对齐模块,并详细描述了实验设置和结果。最后,文章总结了TANGLE的优势,并提出了未来可能的研究方向。
三、利用高斯混合模型构建通用病理幻灯片表示
这篇文章介绍了一种名为PANTHER的无监督学习方法,通过利用组织形态的冗余性,构建了一个紧凑且通用的全幻灯片图像表示,用于计算病理学中的各种临床任务。

一作&通讯
| 作者角色 | 姓名 | 单位名称(英文) | 单位名称(中文) |
|---|---|---|---|
| 第一作者 | Andrew H. Song | Mass General Brigham and Harvard University | 麻省总医院布里格姆和哈佛大学 |
| 通讯作者 | Faisal Mahmood | Mass General Brigham and Harvard University | 麻省总医院布里格姆和哈佛大学 |
这篇文章是关于一种新的无监督学习方法,名为PANTHER(Prototype AggregatioN-based framework for compacT HEterogenous slide set Representation),用于计算病理学中的全幻灯片图像(WSIs)表示学习。
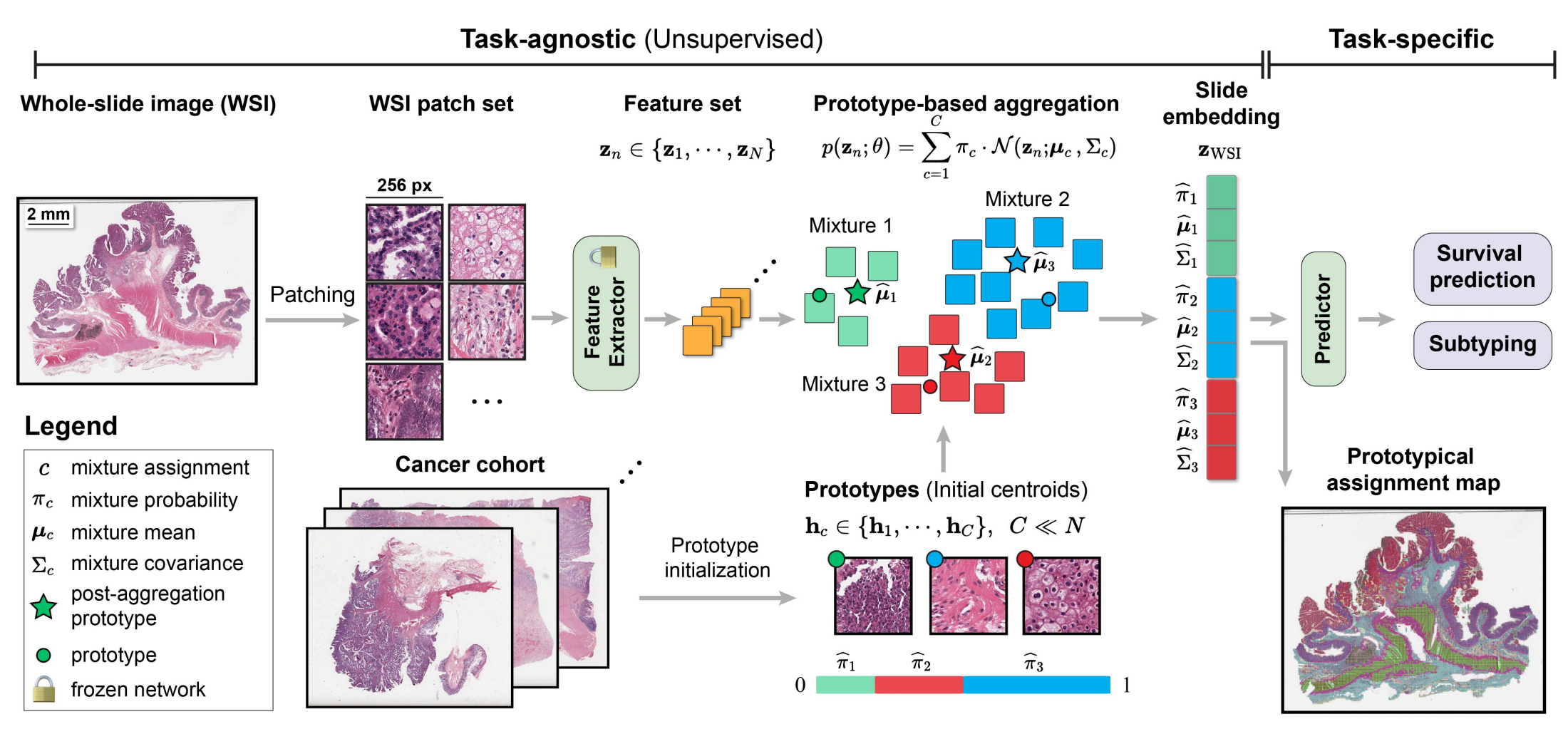
PANTHER基于高斯混合模型(GMM),通过将WSI图像块总结为一组更小的形态原型来构建一个紧凑的幻灯片级表示。这种方法不依赖于特定临床任务的弱监督学习,而是利用组织中的形态冗余来生成通用的幻灯片表示,这有助于在数据有限的情况下提高泛化能力。
文章的主要贡献包括:
- 提出了第一个基于GMM的原型框架,用于学习基于WSI的紧凑和无监督的幻灯片表示。
- 在四个诊断和九个预后任务上对PANTHER进行了全面评估,显示出其性能优于几乎所有无监督和监督基线。
- 通过量化和可视化组织内形态原型的分布,提供了模型可解释性的新视角。
PANTHER通过以下步骤构建幻灯片表示:
- 将WSI分割成小块,使用预训练的视觉编码器提取每个小块的特征嵌入。
- 假设每个小块嵌入是从GMM生成的,每个混合组件代表一个形态学原型。
- 使用期望最大化(EM)算法估计GMM参数,这些参数定义了小块嵌入与原型之间的映射。
- 将估计的GMM参数连接起来形成幻灯片表示,该表示可以用于各种下游任务。
在多个数据集上进行的实验表明,PANTHER在亚型和生存任务上的性能优于或至少与监督MIL基线相当。此外,通过分析形态原型,研究者能够获得有关模型可解释性的新定性和定量见解。
文章还讨论了与PANTHER相关的工作,包括在计算病理学中使用多实例学习(MIL)、度量不同集合间距离的方法,以及基于原型的集合表示学习。此外,文章还介绍了PANTHER的实验设置、数据集、基线比较、实现细节和结果。
最后,文章总结了PANTHER的贡献,并讨论了其局限性,例如对于所有任务使用固定的原型数量C可能不适用于某些癌症类型。
未来的工作可能包括引入更富有表达力的混合模型来描述小块分布,以数据驱动的方式确定原型数量,以及在小样本罕见癌症队列上进行评估。
四、下一代诊断病理技术:AI赋能的多光子显微镜应用与展望
这篇文章综述了人工智能如何赋能无标记多光子显微镜技术,推动病理诊断向更精准、高效的下一代诊断病理发展。

一作&通讯
以下是文章中提到的第一作者和通讯作者,以及他们对应的单位:
| 角色 | 姓名 | 单位名称(中文) |
|---|---|---|
| 第一作者 | 王s | 福州大学机械工程与自动化学院 |
| 通讯作者 | 黄f | 福州大学机械工程与自动化学院 |
| 通讯作者 | 陈ly | 北京大学未来技术学院分子医学研究所 |
| 通讯作者 | 陈jx | 福建师范大学教育部光电子科学与技术重点实验室 |
这篇文章是一篇关于人工智能(AI)在无标记多光子显微镜(MPM)病理诊断中应用的综述。
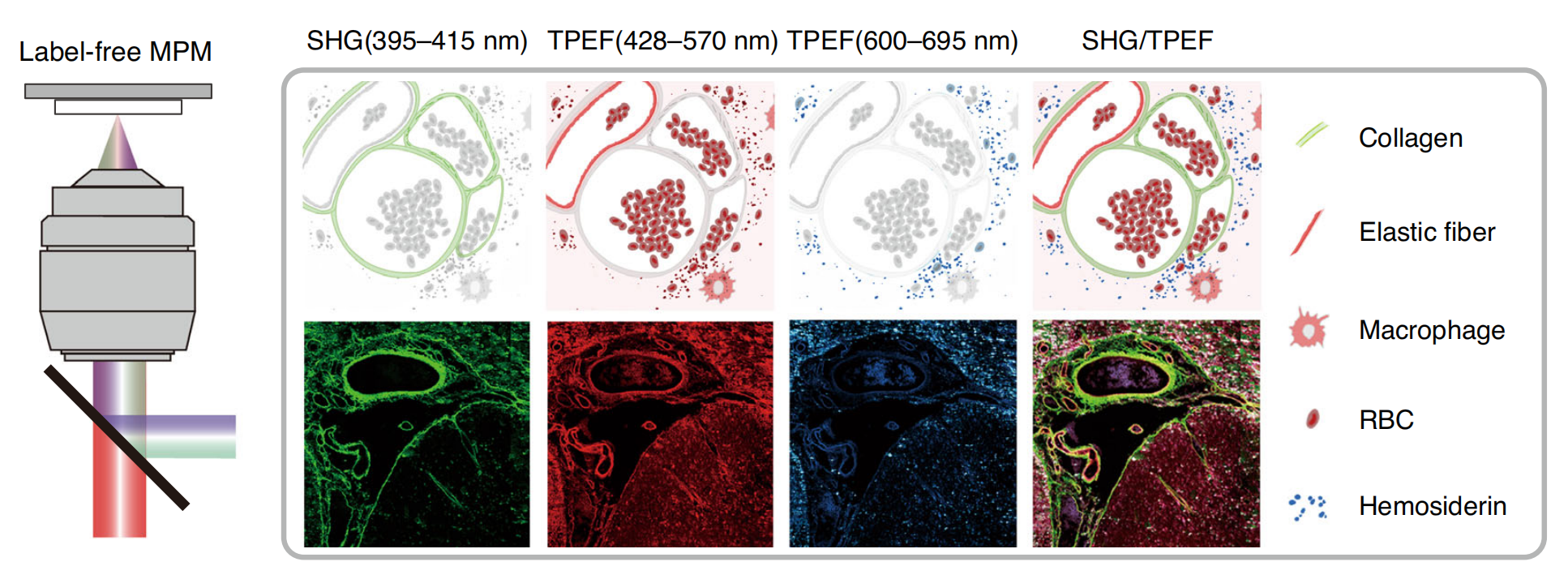
文章首先介绍了传统的病理诊断依赖于病理学家的主观解读,但这种依赖性导致了诊断结果的不一致性。为了提高癌症治疗的精确性,对精确病理诊断的需求不断增加,促使传统病理诊断向“下一代病理诊断”发展,这种诊断方式强调多维度、智能化的诊断方法。
文章详细讨论了多光子显微镜(MPM)在多种人类病理组织中的高分辨率无标记成像能力,以及AI如何提高诊断的准确性和效率。MPM利用与生物组织相互作用的非线性光学效应,能够同时成像生物组织内的多个内在成分。结合AI的MPM进一步提高了诊断的准确性和效率,为基于多光子诊断标准的病理辅助诊断方法提供了希望。
文章系统地概述了MPM在不同人类疾病病理诊断中的应用,并总结了常见的多光子诊断特征。此外,文章还探讨了AI在增强多光子病理诊断中的重要作用,包括图像预处理、精细的鉴别诊断和预后预测等方面。文章还讨论了MPM和AI整合所面临的挑战和前景,包括设备、数据集、分析模型以及与现有临床路径的整合。
最后,文章探讨了AI与无标记MPM之间的协同作用,以构建新的诊断框架,旨在加速智能多光子病理系统在临床设置中的采用和实施。
文章还提到了一些具体的技术细节,例如多光子物理机制、多光子显微镜的仪器发展历史、以及AI在图像处理和诊断中的应用。此外,文章还讨论了多光子显微镜在病理诊断中的一些具体应用案例,以及AI如何辅助病理学家进行更准确的诊断和预后预测。
