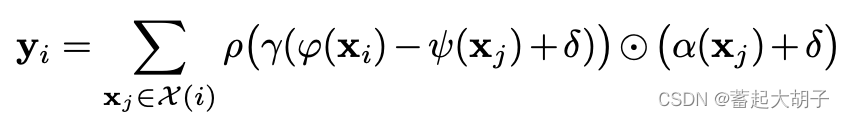基于点云的深度学习方法预测蛋白-配体结合亲和力【Briefings in Bioinformatics, 2022】
论文题目:

研究背景:
1. 三维空间结构及相关信息可以通过深度图像(depth images)、网格(meshes)和体素网格(voxel grids)等多种方式获取、处理、存储和利用。
2. 药物设计领域通常需要准确预测配体(通常是小分子)和靶标蛋白之间的结合亲和力。目前有多种方法可以处理蛋白和配体的3D结构信息,比如:将3D结构信息转化为molecular descriptors、转化为2D的互作图谱、3D体素或图形数据等。但是在本研究中,作者选择了两种基于处理点云数据的神经网络(PointNet和PointTransformer)来构建蛋白-配体结合亲和力预测模型。
3. 点云(point cloud)是由一系列点的三维坐标(x, y, z)构成(有时也会有一些额外的信息,但是坐标信息是必须的),它是一种常用于保存3D对象的原始几何信息的文件格式。
4. PointNet是一种基于DL的处理点云数据的方法,它可以把点的坐标直接作为输入,利用对称函数(symmetry functions)实现置换不变性,通过MLP和Max Pooling对三维坐标信息进行处理。与PointNet相比,PointTransformer则增加了分级的空间结构和自注意力模块。
我的理解是:假设一个三维结构可以由A、B、C、D四个点的坐标来表示,那么按照D,A,C,B的顺序来存储这四个点的坐标并不会影响该三维结构。在这种情况下,用对称函数处理坐标信息也不会受到坐标的先后顺序的影响。比如说:max([A,B,C,D]),无论怎样改变ABCD的顺序,最终的值都是这四个值中最大的那一个。
可以参考B站的这个视频https://www.bilibili.com/video/BV1Sv4y1S7x7/?share_source=copy_web&vd_source=9f1b2c7e5841d2b27667d708de6a93d0
5. 利用PointNet处理点云数据可以极大减少预处理的时间,同时保持结构信息的相对完整。
材料和方法:
首先明确这是一个回归任务,所以作者用相关性系数来评估最终结果。
数据集:
关于PDBbind-2016数据集,可见:
https://blog.csdn.net/2201_75349501/article/details/128225366
数据预处理:
1). 去掉配体为多肽的复合物(590);
2). 去掉共价连接的配体的复合物(379);
3). 去掉配体不完整的复合物(481);
数据集划分:
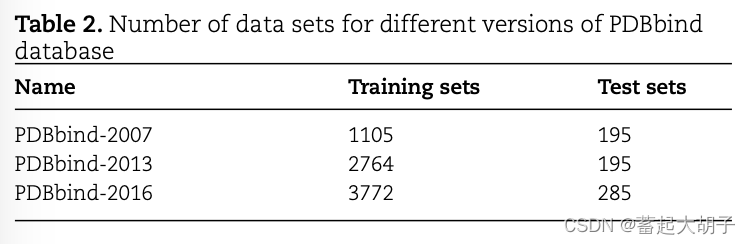
训练集:PDBbind-2016 的 refined set(包括3772个protein-ligand complex data sets);
测试集:PDBbind-2016 的 core set(包括285个protein-ligand complex data sets,从训练集中去掉所有测试集的数据);
拓展数据集:为了评估模型对于更大数据集的预测效果, 整合 PDBbind-2016 的 refined 和 general sets(包括11327个protein-ligand complex data sets);
此外,还有PDBbind-2007和PDBbind-2013的数据集,如下所示:
数据集划分后的处理:
1). 将数据集中的溶剂分子、金属离子等去除,结合亲和力取负对数,之后转化为点云数据格式;
2). 为了评估不同原子属性对于预测结果的影响,额外增加三种不同的特征用于模型训练:
I. 七种原子类型:hydrogen, carbon, nitrogen, oxygen, phosphorus, sulfur and halogen(氢,碳,氮,氧,磷,硫,卤);
II. 原子的采样数目由1024增加到2048;
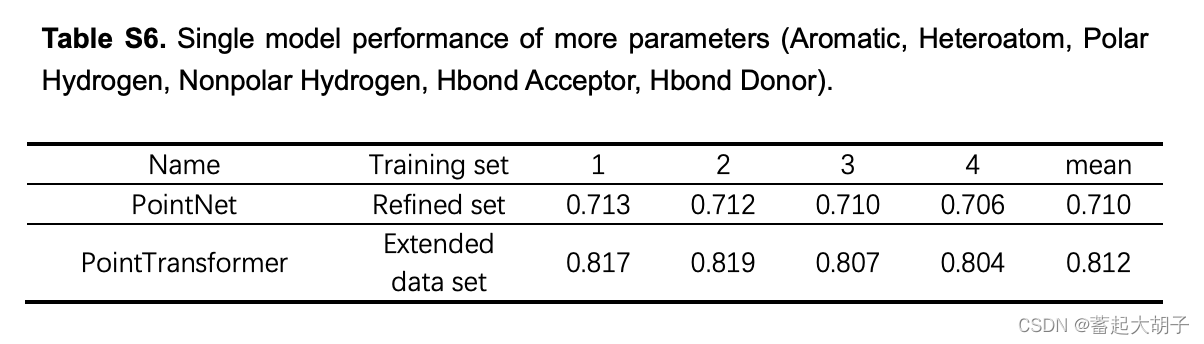
III. 训练集和拓展数据集中,每个原子的理化性质(包括:芳族,杂原子,极性氢,非极性氢,氢键受体和氢键给体);
思路:
1. 在合并点云进行训练之前,我们将蛋白质-配体复合物的坐标对齐到配体中心,确保模型不受平移的影响。
2. 收集配体的所有原子以及与配体对应的蛋白上的外层原子,一共1024个点。为了简化计算,将这些点看作独立的点,不考虑配位键的问题。
3. 每个原子都由6个数据构成(x, y, z coordinates, van der Waals radius, atomic weight, source [1 for protein; -1 for ligands])。原子坐标由距离配体中心最远的原子距离做标准化处理,van der Waals redius和atomic weight也被另外进行标准化处理。
预测流程:
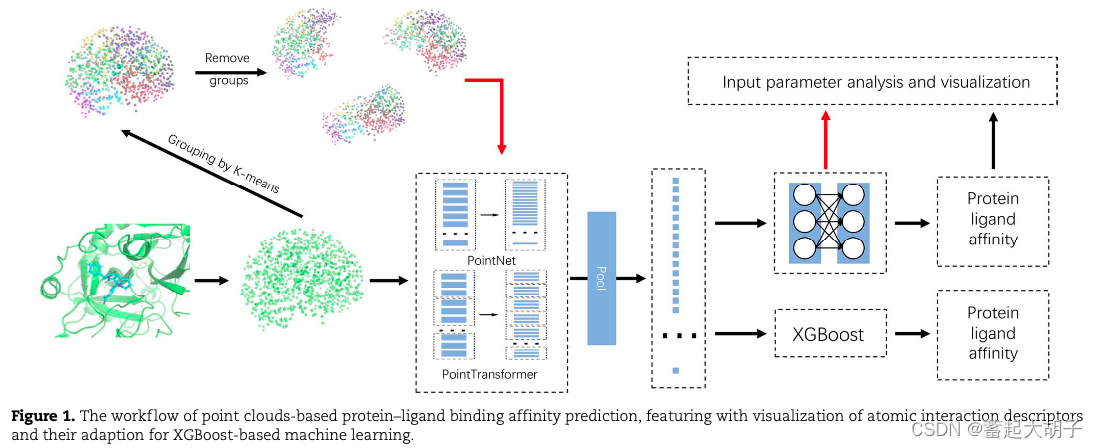
整个过程分为两条线:预测protein-ligand结合亲和力 & protein-ligand 互作特征可视化。
可视化:用K-means将1024个point聚为20个groups,然后独立删掉每一个group,用PointNet和PointTransformer预测结合亲和力,记录下预测结果和实际结果之间的差值。如果差值>均值,那么认为这个group是一个能够影响结果的关键的group。当差值>=均值,标记为红色;否则标记为蓝色(这个和流程图中那些多种颜色之间没啥关系,流程图中是cluster之后的结果,后面的红蓝标记结果在附件中)。
由于不太关注可视化方面,所以该流程中的可视化部分没有细看。
protein-ligand结合亲和力预测:
point cloud --> PointNet OR PointTransformer --> Pool --> XGBoost --> Protein-ligand affinity
该部分的详细信息如下所述。
模型框架:
1. PointNet框架:
参考: https://doi.org/10.48550/arXiv.1612.00593
https://www.bilibili.com/video/BV1Sv4y1S7x7/?share_source=copy_web&vd_source=9f1b2c7e5841d2b27667d708de6a93d0
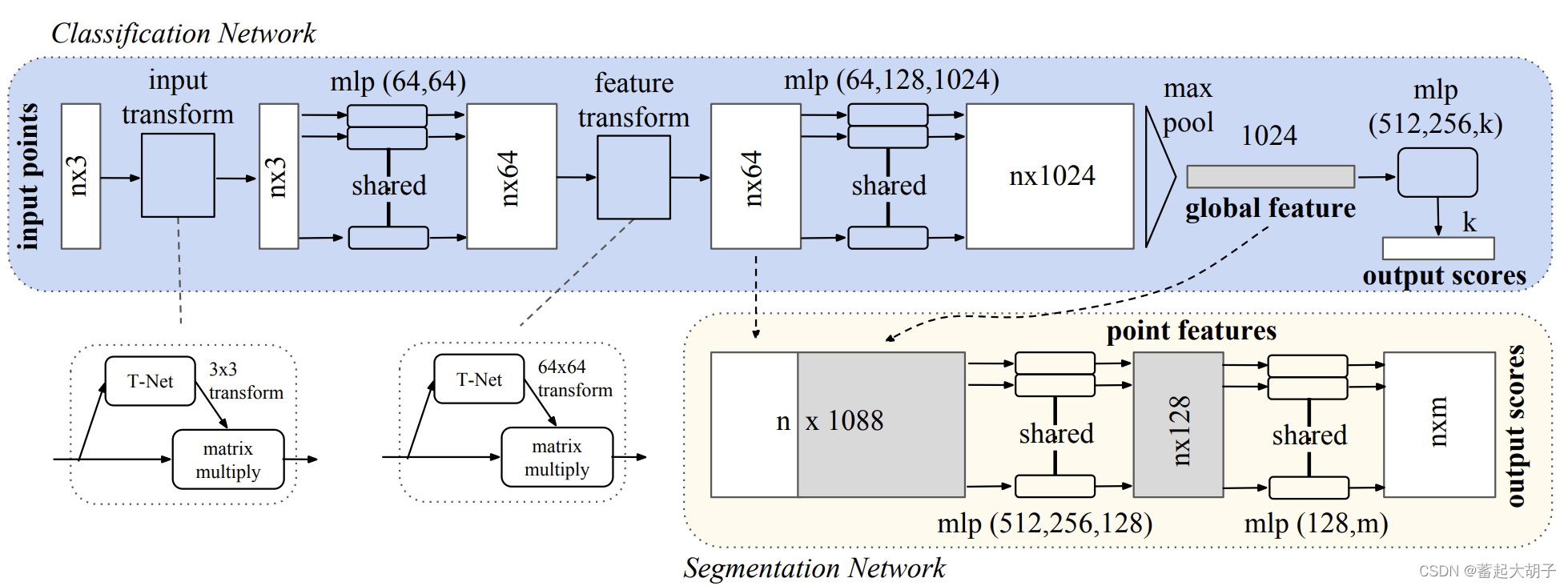
1.1 原始的 PointNet 框架:
原始的PointNet框架如上图所示:可以分为 Classification Network 和 Segmentation Network 两部分。
1). Classification Network
input points 就是point cloud中的原子坐标(如果有法向量,那么就是 nx6);
input points 经过 T-Net (3x3的transform),二者相乘,得到nx3;MLP升维,得到nx64;T-Net (64x64的tranform)相乘,得到nx64; MLP升维,得到nx1024;max pool,得到 1x1024 的global feature (max pool是对nx1024中按照列取最大值,即每一列保留一个最大值,所以最终得到1x1024的矩阵作为全局特征);再经过MLP进行降维,得到 1xk 的output scores。
2). Segmentation Network
前面都一样,只是在后面得到全局特征(global feature)后,将global feature矩阵(1x1024)与前面的nx64的局部特征矩阵相拼接,得到 nx1088(这里的global feature 会进行广播,将1x1024转变为nx1024);之后经过MLP降维得到nx258(点的特征 point features,相当于全局特征&局部特征);再经过MLP得到nxm的output scores。
这里用MLP对输入特征进行升维,目的是产生更多的冗余信息,从而在Max Pooling操作时尽可能减少信息损失
global feature是对nx1024矩阵取Max Pooling后的结果,也就是说每一列只保留一个最大值,最终得到1x1024的矩阵。之所以称之为global feature,我觉得在Max Pooling过程中,每个特征只保留了一个位置上的原子对应的最大值,所以最终得到的是由全局的最大值构成的矩阵,也就是全局的特征。
point features是将全局特征与所有特征的整合,经降维之后仍旧保留了n个点,即得到的是每个点的特征(这个point feature既包含所有特征,也包含全局特征)。
因为Classification Network需要对3D对象进行分类,所以只需要全局的特征即可;而Segmentation Network需要对3D对象进行分割,所以需要保留全局和局部特征。
T-Net 结构:
参考:https://github.com/yanx27/Pointnet_Pointnet2_pytorch/blob/master/models/pointnet_utils.py
3x3和64x64的结构是一样的,3x3的T-Net的目的在于对 input 进行 automatic alignment;
64x64的T-Net的目的在于对 Embedding Space Alignment;
class STN3d(nn.Module):
def __init__(self, channel):
super(STN3d, self).__init__()
self.conv1 = torch.nn.Conv1d(channel, 64, 1) ## 输入特征数母、输出特征数目、卷积核
self.conv2 = torch.nn.Conv1d(64, 128, 1)
self.conv3 = torch.nn.Conv1d(128, 1024, 1)
self.fc1 = nn.Linear(1024, 512)
self.fc2 = nn.Linear(512, 256)
self.fc3 = nn.Linear(256, 9)
self.relu = nn.ReLU()
self.bn1 = nn.BatchNorm1d(64) ## 批标准化
self.bn2 = nn.BatchNorm1d(128)
self.bn3 = nn.BatchNorm1d(1024)
self.bn4 = nn.BatchNorm1d(512)
self.bn5 = nn.BatchNorm1d(256)
def forward(self, x):
batchsize = x.size()[0] ## x的构成:batchsize, 3维(x,y,z)和6维(x,y,z + 法向量), 1024(取得点的数目) = x.size()[0], x.size()[1], x.size()[2]
x = self.relu(self.bn1(self.conv1(x)))
x = self.relu(self.bn2(self.conv2(x)))
x = self.relu(self.bn3(self.conv3(x)))
x = torch.max(x, 2, keepdim=True)[0] ## 按照dim=2取最大值,保留维度,[0]是取 values 的张量
x = x.view(-1, 1024) ## 将 x 的 shape 改成 1x1024 (x.view(-1,1024)生成1x1024;如果是x.view(-1,512)的话,则生成2x512)
x = self.relu(self.bn4(self.fc1(x)))
x = self.relu(self.bn5(self.fc2(x)))
x = self.fc3(x)
iden = Variable(torch.from_numpy(np.array([1, 0, 0, 0, 1, 0, 0, 0, 1]).astype(np.float32))).view(1, 9).repeat(
batchsize, 1) ## 创建一个 batchsize x 1 x 9 的 Tensor,其 value 是 [1, 0, 0, 0, 1, 0, 0, 0, 1]。这一步的目的是创建一个初始化的单位矩阵(见 https://arxiv.org/pdf/1612.00593.pdf)
if x.is_cuda:
iden = iden.cuda()
x = x + iden ## affine transformation:“仿射变换”就是一种简单的变换,它的变化包括旋转、平移、伸缩,原来的直线仿射变换后还是直线,原来的平行线经过仿射变换之后还是平行线,这就是仿射。
x = x.view(-1, 3, 3) ## 生成 batch x 3 x 3
return x
整体上来看,正如作者所言,T-Net 就像是一个 mini 的 PointNet。其结构和PointNet的结构是比较相近的,T-Net的目的是对特征进行仿射变换。我觉得做仿射变换的目的在于将空间坐标进行平移、旋转这些操作,从而增强模型的泛化能力(自己想的,不一定对)。
1.2 本文中的 PointNet 框架:
参考:本文附件
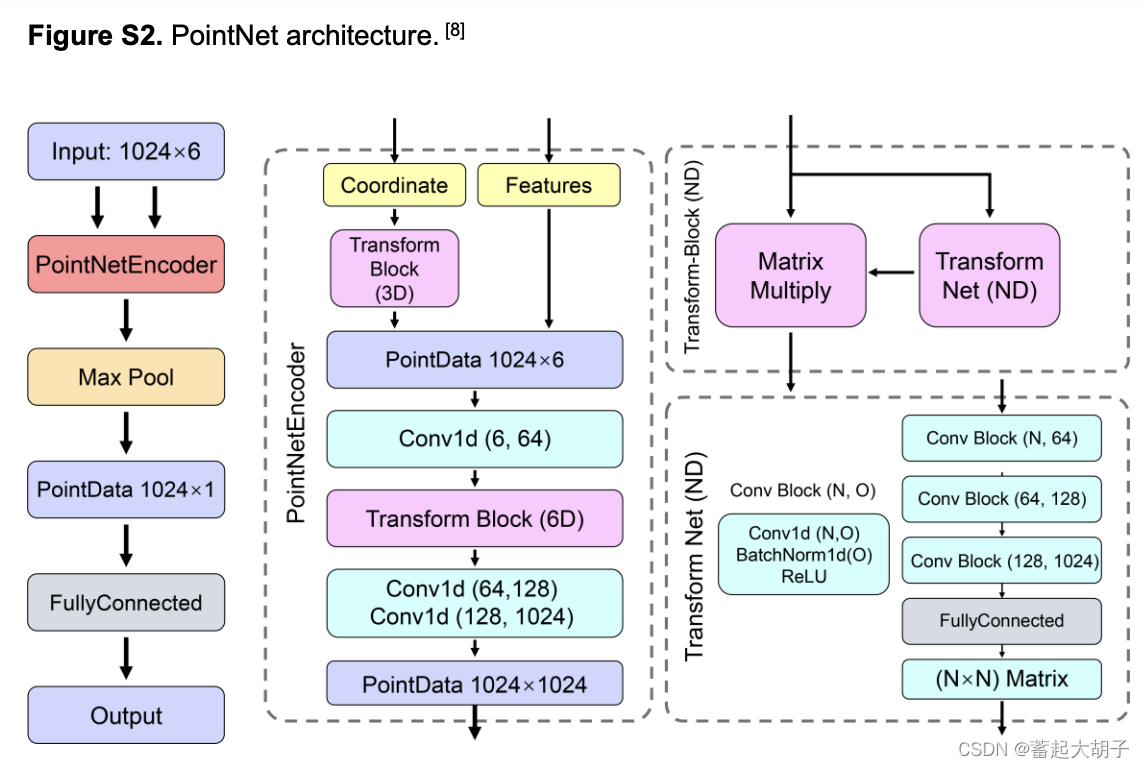
和上面原始的PointNet框架基本一致,只不过这里除了坐标(Coordinate)外,还加了一些额外的特征(Features)。
在对坐标进行Transform Block(3D)处理之后,与Features矩阵拼接并升维之后,再用Transform Block(6D)处理一次,经过升维完成对PointNetEncoder。按照原始的PointNet框架,可以完全直接将这6个特征整合一起作为原始输入,但作者这里单独对坐标进行transform操作
原因我觉得可能是:
原始的PointNet框架输入6个特征的话,那这6个特征包括3个坐标及对应的法向量,也就是说这6个特征都是和坐标相关的;而本文作者这里的6个特征中,3个是坐标,剩下的3个则是原子的理化性质(范德华半径、原子质量和原子来源),也就是说这6个特征不是同一个属性的,所以需要分开处理。
本文PointNet框架和原始的PointNet框架的代码基本是一致的。
2. PointTransformer 框架:
2.1 原始的 PointTransformer 框架:
参考: https://doi.org/10.48550/arXiv.2012.09164
https://github.com/qq456cvb/Point-Transformers
https://zhuanlan.zhihu.com/p/234638980

如 Figure 3 所示,PointTransformer 也可以分为 segmentation 和 classification 两部分。
segmentation 部分:主要是降采样 Point Transformer和升采样 Point Transformer的过程。Transition Down & Point Transformer 对特征进行降采样,得到 point-wise 的特征,最终得到的 N/256 x 512 可以被视为 global feature。接着通过 Transition Up & Point Transformer 以及 skip connection 实现上采样并得到 global + local 的 point-wise 特征,使最终得到的 point-wise 特征兼具全局和局部特征,使之更具有区分度,从而实现对3D结构的语义分割。
classification 部分:基本上就是segmentation前半部分,通过降采样得到全局特征,之后再经过 Global AvgPooling 和 MLP 实现对3D结构的分类。
整体上给我的感觉就是:PointTransformer 和 PointNet++ 很像(可见:https://zhuanlan.zhihu.com/p/266324173)。
具体的每个子模块的结构:
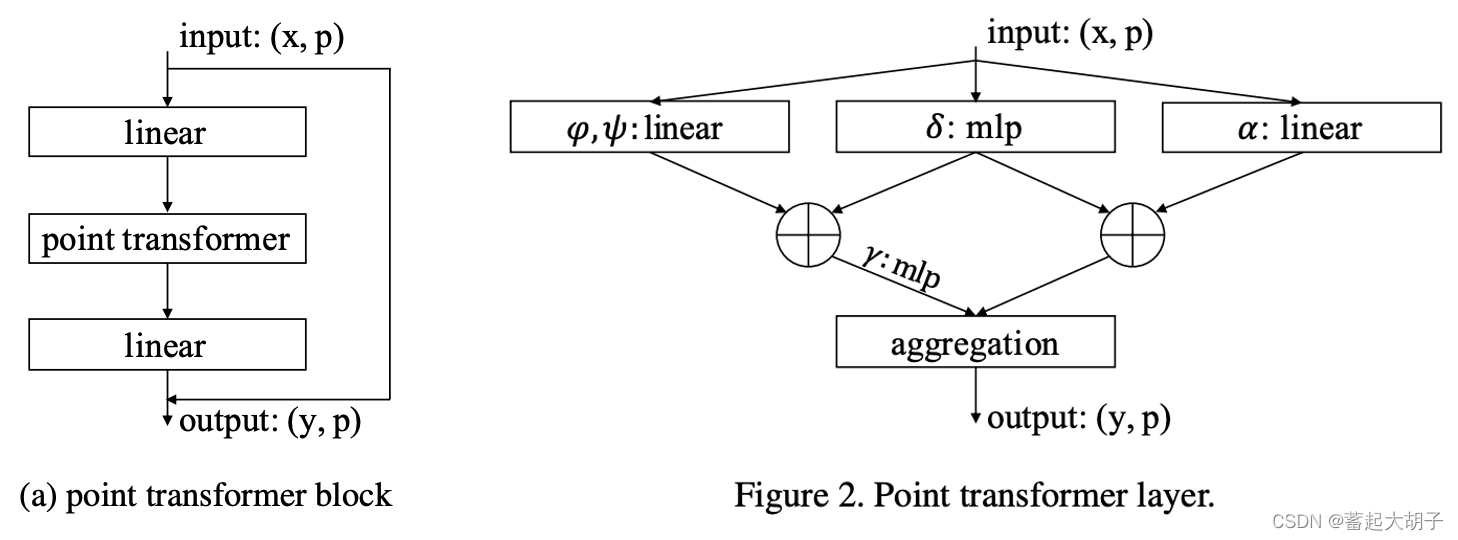
1). point transformer block:
公式:
框架:
x: a set of feature vectors;
p: 3D coordinates
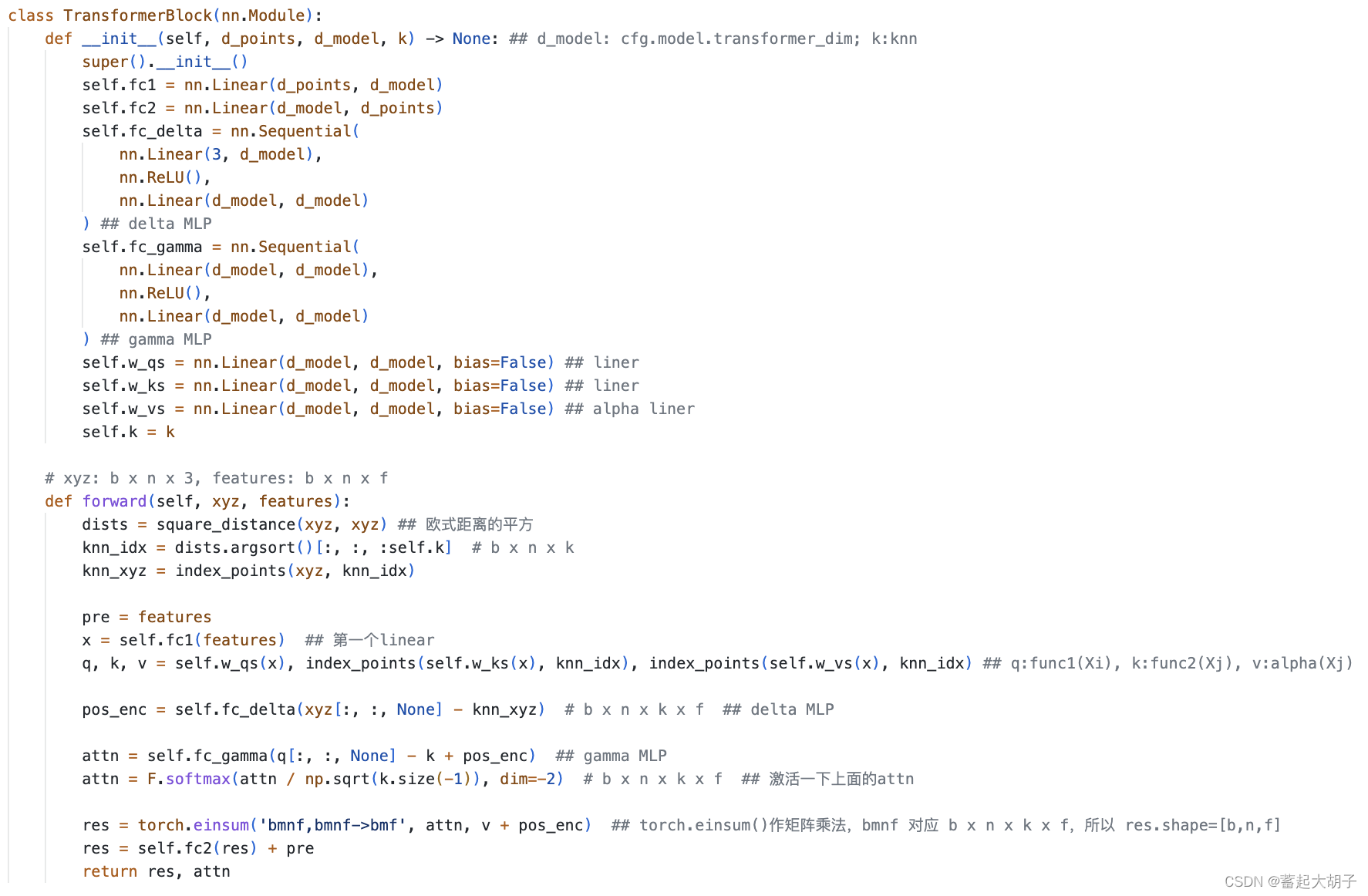
代码(可见https://github.com/qq456cvb/Point-Transformers/blob/master/models/Hengshuang/transformer.py):
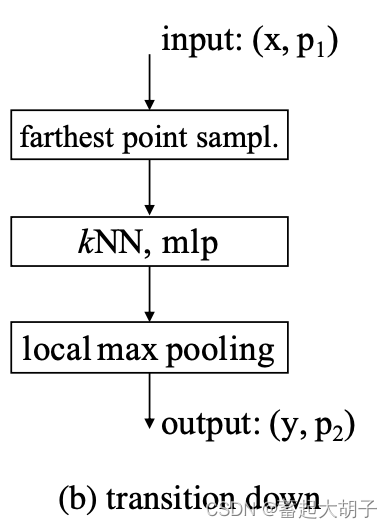
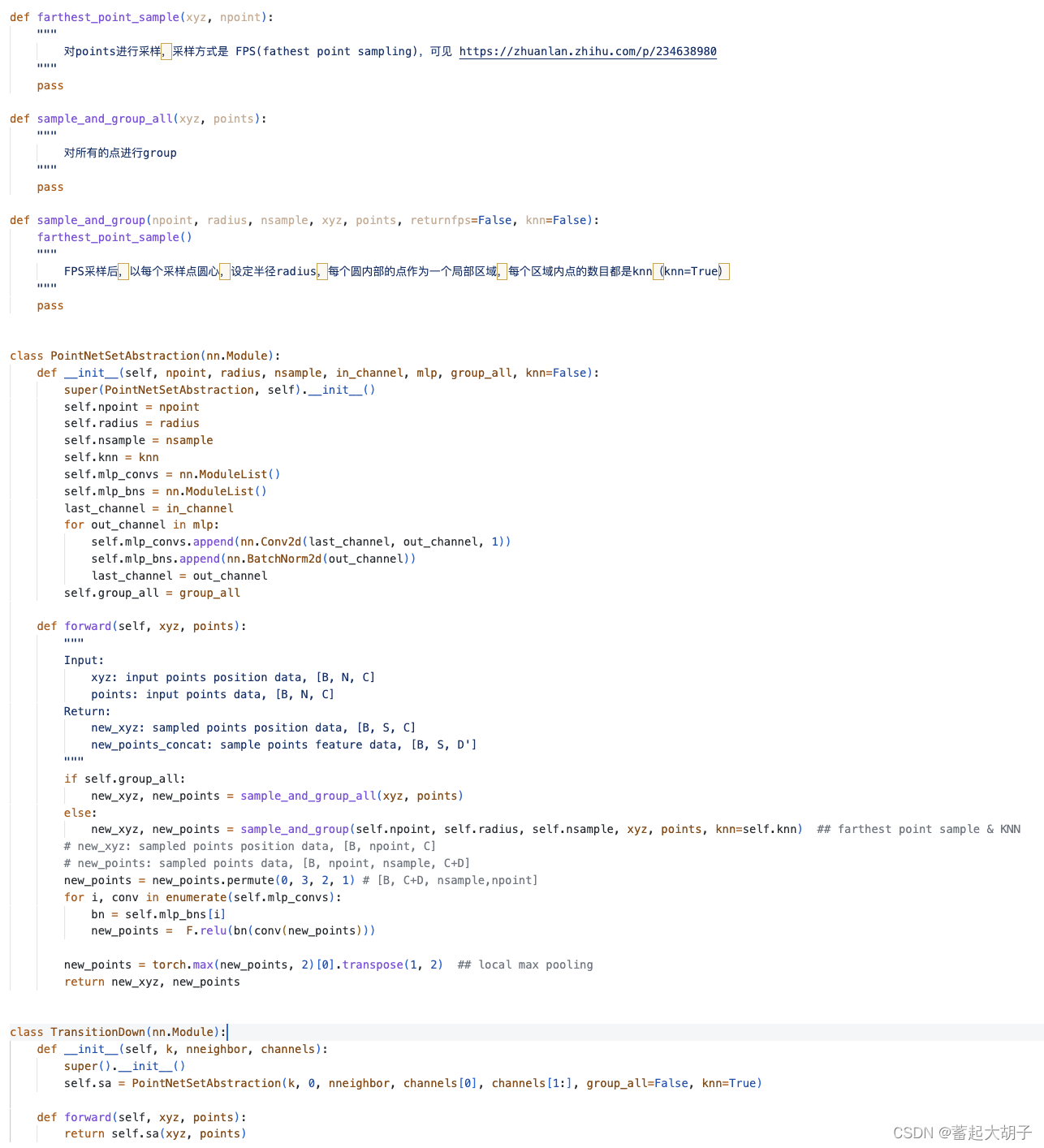
2). Transition Down(下采样):
框架:
代码(可见 https://github.com/qq456cvb/Point-Transformers/blob/master/models/Hengshuang/model.py):
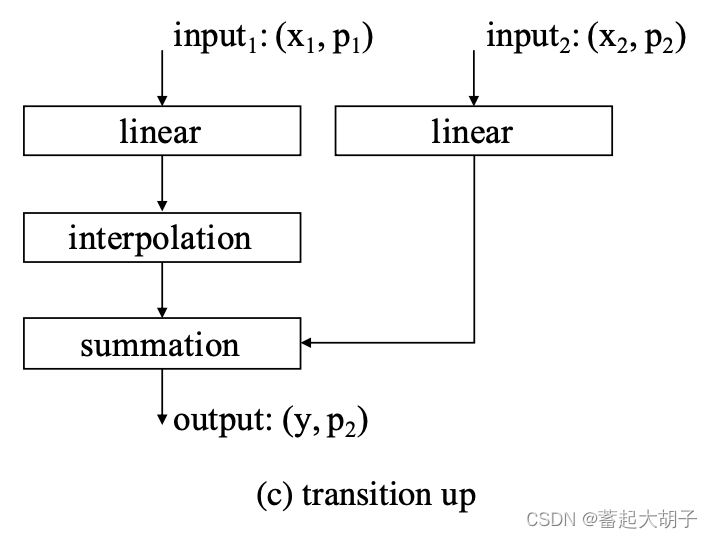
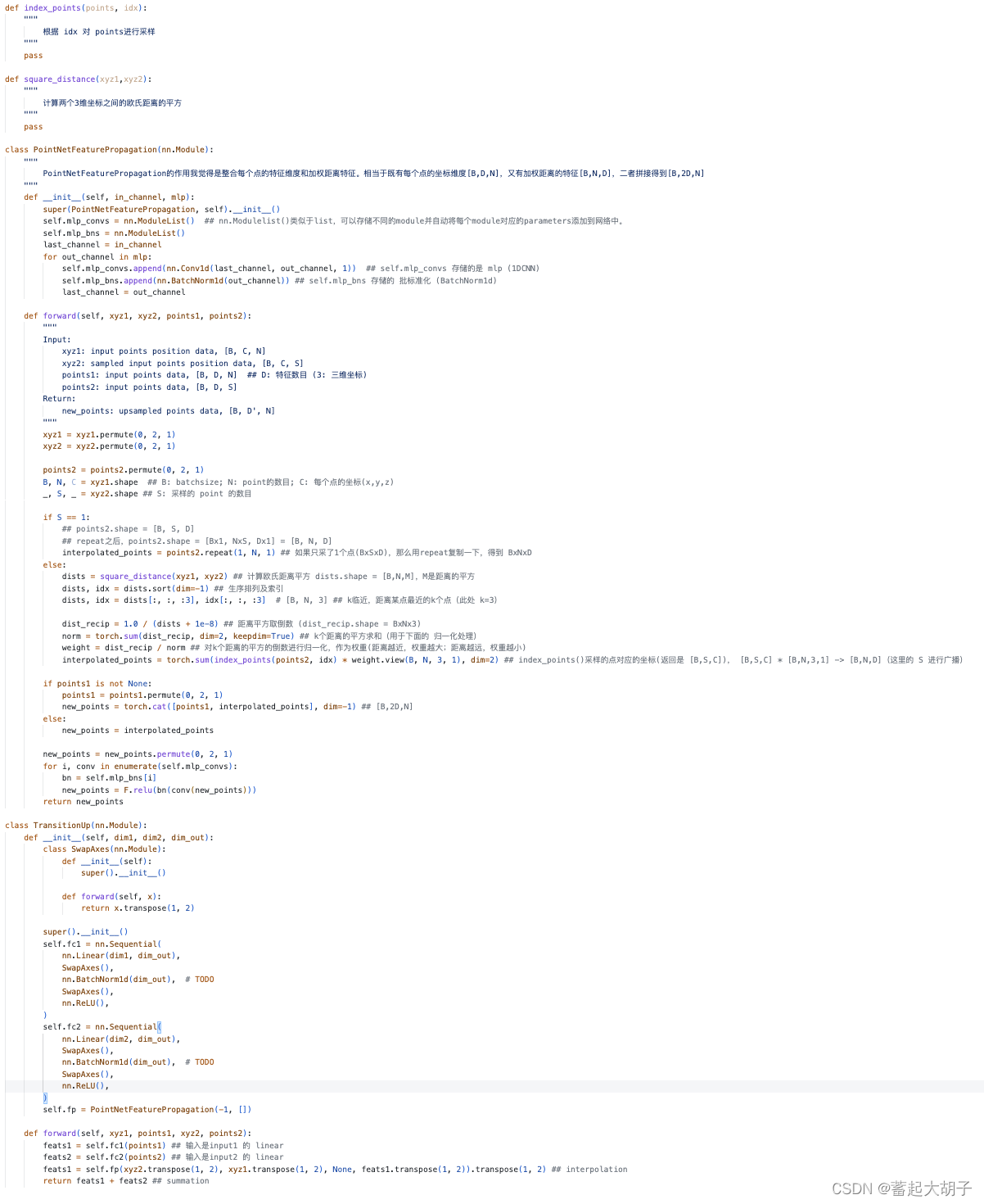
3). Transition Up(上采样):
框架:
代码(可见 https://github.com/qq456cvb/Point-Transformers/blob/master/models/Hengshuang/model.py):
2.2 本文中的 PointTransformer 框架:
参考:本文附件
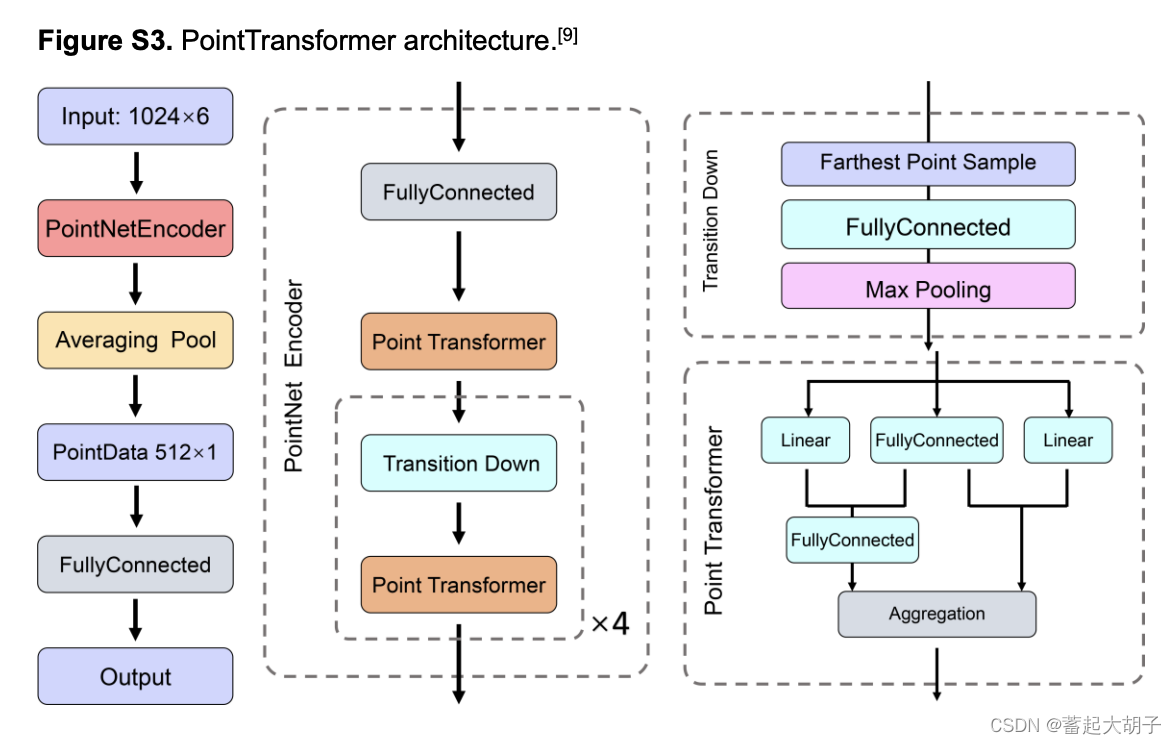
本文中的 PointTransformer 和 原始的 PointTransformer 框架中的 classification 的结构是一致的(代码基本也是一样的)。
模型预测效果:
作者分别用PDBbind-2016 refined set 和 Larger data sets 构建模型,然后在CASF-2016标准数据集上进行测试,结果如下:
PointNet-1024 (R)和PointNet-AtomChannel (R)相比,增加额外原子类型特征可以提高Person’s correlation coefficient R,降低 MAE 和 RMSE。PointNet-1024 (R)和PoingNet-2048 (R)相比,增加采样点的数目,也可以提升模型的性能。
PointNet (AtomChannel) with seven additional channels including hydrogen, carbon, nitrogen, oxygen, phosphorus, sulfur and halogen.
-
PointTransformer (1024)和PointNet-1024相比,预测效果差别不大。 -
整体上
PointNet或PointTransformer在PDBbind-2016 refined set上的效果不如PSH-GBT和TopBP(complex)。
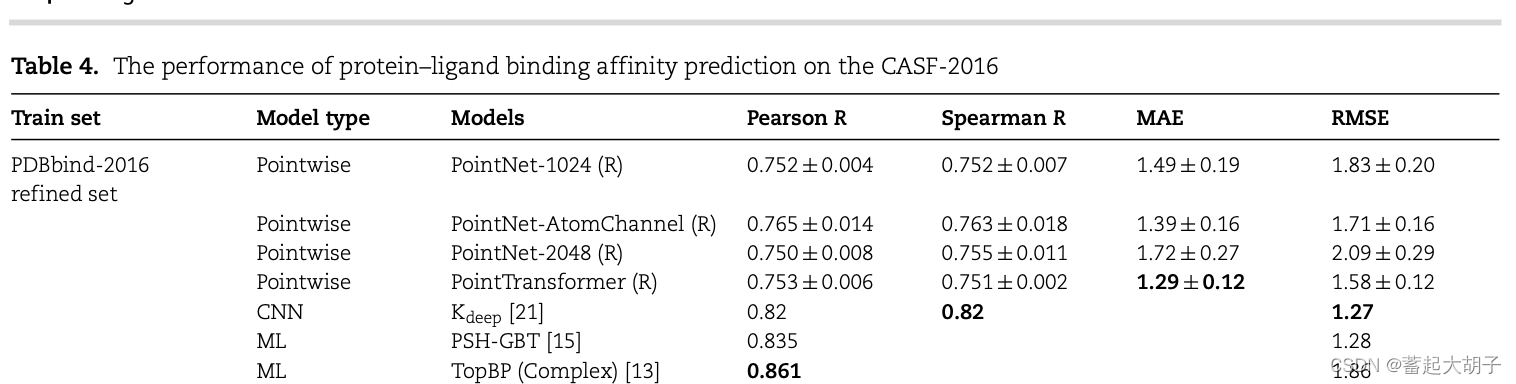
-
但是增加额外的原子理化性质特征反而会降低模型的性能。
原因作者认为在点云数据中添加额外的原子理化性质可能会使模型混淆对原子性质的理解。
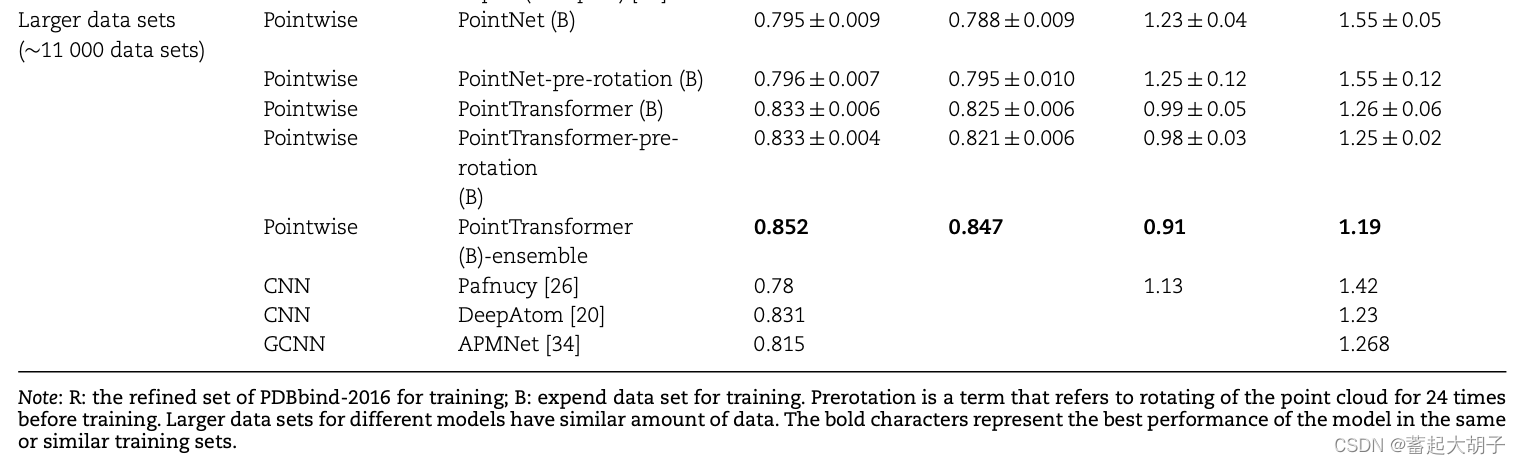
- 在更大的数据集上测试发现,数据集越大,模型效果越好;训练之前对点云坐标进行旋转(rotating of the point cloud for 24 times before training)可以提升模型预测效果;将多个模型进行整合(5个最好的模型整合为
PointTransformer-ensemble)可以进一步提升模型性能。
坐标旋转:将训练集和测试集中的point cloud绕配体的中心沿x、y、z旋转0,90,180和270度,将所有结果取均值作为该坐标旋转之后的结果。
5个最好的模型见附件。
总结:
- 基于结构特征的DL模型对于protein-ligand亲和力的预测表现出巨大的潜力;
- 与其他ML或DL方法相比,基于点云数据的模型减少了数据预处理的耗时,测试结果也表明基于点云数据的PointNet和PointTransformer在protein-ligand亲和力预测方法表现突出,可以促进protein-ligand互作预测方法的发展;
- 与PointNet相比,PointTransformer更加耗时,所以优化模型结构有可能提升模型的性能;
- PointNet可以被用做 protein-ligand complex 的编码器,从而使点云数据得到更广泛的应用(比如用于强化学习);同时也可以开发一些应用于其他方面的模型,比如酶活性位点的研究、分子对接等;